tasiРНК
Транс-активи́рующие ма́лые интерфери́рующие РНК, tasiРНК, TAS РНК[1] (англ. trans-acting siRNA) — группа малых некодирующих РНК наземных растений, подавляющие экспрессию генов путём пост-трансляционного сайленсинга[2][3][4]. TasiРНК транскрибируются в геноме в форме двуцепочечных полиаденилированных РНК, которые в дальнейшем процессируются и превращаются во фрагменты РНК длиной 21 нуклеотид[2]. Эти фрагменты включаются в РНК-индуцируемый комплекс выключения гена (RISC). tasiРНК часто относят к малым интерферирующим РНК (siРНК) ввиду того, что обе этих группы малых РНК транскрибируются в форме двуцепочечных РНК и подвергаются схожему процессингу. Впрочем, tasiРНК отличаются от других siРНК тем, что они связывают свои последовательности-мишени с меньшей специфичностью[3]. В этом их механизм более схож с механизмом действия микроРНК, так как они не нуждаются в полной комплементарности последовательностей со своей мишенью, чтобы направлять её распад[5].
История открытия
Существование tasiРНК впервые было установлено в 2004 году двумя группами учёных, работавших с арабидопсисом. Обе статьи были опубликованы в октябре того года с разницей в несколько дней. Первая группа (Peragine с сотрудниками) изучала белок ZIPPY (ZIP) из группы Argonaute, а вторая (Vazquez с сотрудниками) пыталась найти специфичные siРНК. Несмотря на то, что группы начали с разных стартовых точек, обе по ходу исследований сфокусировались на изучении специфичного растительного белка супрессора сайленсинга генов 3 (SGS3) и фермента РНК-зависимой РНК-полимеразы 6 (RDR6). Обе группы пришли к заключению, что эти белки играют важную роль в образовании специфических siРНК — tasiРНК[2][3].
Из-за ключевых отличий, отделяющих tasiРНК от остальных групп некодирующих РНК, tasiРНК были новой открытой группой РНК, хотя они имеют общие черты с siРНК и микроРНК. В отличие от микроРНК, tasiРНК образуются из длинных двуцепочечных РНК, и их образование зависит от RDR6. От siРНК tasiРНК отличаются тем, что они вызывают разрушение транскриптов с различающимися последовательностями. В этом отношении tasiРНК схожи с микроРНК, однако механизм их процессинга сближает из с siРНК[5].
Образование
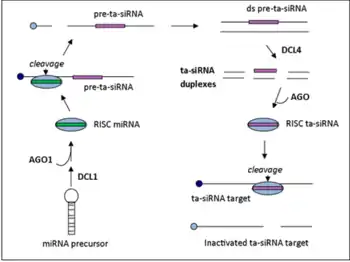
TasiРНК образуются из длинных некодирующих транскриптов с помощью разрезания их белками Argonaute, направляемого микроРНК. Этот путь включает превращение одноцепочечного разрезанного транскрипта в двуцепочечный при помощи RDR6 и SGS3[6]. Образующаяся двуцепочечная РНК разрезается Dicer-подобным ферментом 4 (DCL4) (гомолог Dicer животных[1]) с образованием коротких фрагментов РНК длиной 21 нуклеотид, которые и становятся tasiРНК[7][8].
У Arabidopsis thaliana имеются 4 локусов и групп локусов, кодирующих tasiРНК. Для процессинга продуктов генов TAS1, TAS2, и TAS4 необходим один сайт связывания микроРНК, а для процессинга продуктов TAS3 необходимо два сайта связывания микроРНК[9]. Гены TAS у разных растений не являются ортологами, то есть семейство генов TAS1 у мха не имеет общего гена-предка с TAS1 арабидопсиса. Среди TAS1 выделяют TAS1a, TAS1b и TAS1c, эти три локуса являются паралогами и имеют определённое сходство с TAS2, так что, по-видимому, все четыре локуса — паралоги. На то, что транскрипты этих генов вряд ли кодируют белок, указывает то, что эти транскрипты не имеют протяжённых открытых рамок считывания и могут кодировать пептиды длиной не более 50 аминокислотных остатков[1].
Все гены tasiРНК имеют два экзона, причём в случае TAS1 и TAS2 сайт разрезания микроРНК находится в интроне, так что процессингу с помощью RDR6 подвергаются несплайсированные предшественники. Хотя почти все найденные tasiРНК соответствуют фрагментам транскрипта-предшественника, для каждого из них была найдена хотя бы одна tasi-РНК, соответствующая минус-цепи. Такие tasiРНК, видимо, могут регулировать содержание в клетке собственного предшественника[1].
TAS1 и TAS2
Транскрипты TAS1/2 подвергаются первичному процессингу в форме AGO1-опосредованного разрезания по 5'-концам, направляемого miR173. После этого RDR6 переводит транскрипт в двуцепочечную форму, которая далее процессируется DCL4 с образованием tasiРНК длиной 21 нуклеотид. Эти tasiРНК связываются с мРНК-мишенями за счёт двух выступающих нуклеотидов на 3'-конце, действуя, таким образом, как транс-регуляторные элементы[9].
TAS4
Начальные этапы процессинга транскриптов TAS4 схожи с таковыми у TAS1/2. Вначале они подвергаются AGO1-опосредованному разрезанию, направляемому miR828, далее происходит образование двуцепочечных РНК и процессинг с помощью DCL4[9].
TAS3
В отличие от TAS1/2 и TAS4, для процессинга TAS3 необходимы два сайта связывания с микроРНК (miR390). Транскрипт сначала разрезается по 3'-сайту связывания при помощи AGO7. Далее, как и в случае TAS1/2 и TAS4, RDR6 синтезирует вторую цепь РНК и образующаяся двуцепочечная РНК далее процессируется DCL4[9].
Механизм
Эндогенные tasiРНК действуют посредством гетеросайленсинга, то есть те гены-мишени, которые tasiРНК репрессируют, не имеют значительного сходства с генами, с которых транскрибируются эти tasiРНК. Это обстоятельство отличает tasiРНК от siРНК, которые осуществляют аутосайленсинг и подавляют экспрессию генов, которые имеют последовательности, идентичные или очень похожие на последовательности генов, от которых siРНК происходят. До открытия tasiРНК считалось, что только микроРНК способны к гетеросайленсингу[2]. Как и siРНК, tasiРНК включаются в состав комплекса RISC, где они направляют комплекс к разрезанию мРНК-мишени в середине сайта комплементарного связывания, тем самым подавляя трансляцию[2][3][10].
Белки группы Argonaute включаются в состав комплексов, осуществляющих сайленсинг генов посредством РНК, в том числе и RISC, который катализирует разрушение мРНК[10][11]. В частности, у арабидопсиса AGO7/ZIPPY участвует в tasiРНК-направляемой регуляции, причём tasiРНК транскрибируются с TAS3. AGO7/ZIPPY связывается с tasiРНК, произошедшими от TAS3, и приступает к разрушению мишеней. По-видимому, AGO7/ZIPPY не взаимодействует с tasiРНК, транскрибированных с TAS1 и TAS2, так что у арабидопсис различные семейства tasiРНК действуют слегка по-разному[11]. У арабидопсиса tasiРНК могут связываться не только с AGO7, но и с AGO1 и также направлять распад мРНК-мишени[12].
Распространение и функции
TasiРНК были обнаружены не только у арабидопсиса[8], но также у мха Physcomitrella patens[6], кукурузы[13] и риса посевного[14]. Примером tasiРНК, выявленной не только у арабидопсиса, но и у всех вышеперечисленных растений, может служить tasi-РНК фактор ответа на ауксин (tasiR-ARF) (группа TAS3). TasiR-ARF вовлечен в сигнальные пути фитогормона ауксина, вызывая разрушение мРНК-мишеней, которые кодируют несколько факторов ответа на ауксин (ARF)[13]: ARF2, ARF3 и ARF4[1]. Показано, что нарушение образования этих tasiРНК приводит к фенотипическим нарушениям. Мишенями других tasiРНК служат несколько генов с не идентифицированной функцией[1].
Показано, что из транскриптов TAS1a и TAS2 получаются не только РНК длиной 21 н., но также несколько tasiРНК длиной 24 н. Точные мишени этих РНК не установлены, но известно, что у растений малые РНК такой длины вовлечены в транскрипционный сайленсинг. Для образования tasiРНК длиной 24 н., похоже, используется альтернативный путь: для их процессинга не нужно участия микроРНК и необходим другой гомолог Dicer животных — DCL3[1].
Примечания
- Макарова Ю. А., Крамеров Д. А. Некодирующие РНК (рус.) // Биохимия. — 2007. — Т. 72, № 11. — С. 1427—1448. Архивировано 14 июля 2014 года.
- Vazquez F., Vaucheret H., Rajagopalan R., Lepers C., Gasciolli V., Mallory A. C., Hilbert J. L., Bartel D. P., Crété P. Endogenous trans-acting siRNAs regulate the accumulation of Arabidopsis mRNAs. (англ.) // Molecular cell. — 2004. — Vol. 16, no. 1. — P. 69—79. — doi:10.1016/j.molcel.2004.09.028. — PMID 15469823.
- Peragine A., Yoshikawa M., Wu G., Albrecht H. L., Poethig R. S. SGS3 and SGS2/SDE1/RDR6 are required for juvenile development and the production of trans-acting siRNAs in Arabidopsis. (англ.) // Genes & development. — 2004. — Vol. 18, no. 19. — P. 2368—2379. — doi:10.1101/gad.1231804. — PMID 15466488.
- Axtell M. J., Jan C., Rajagopalan R., Bartel D. P. A two-hit trigger for siRNA biogenesis in plants. (англ.) // Cell. — 2006. — Vol. 127, no. 3. — P. 565—577. — doi:10.1016/j.cell.2006.09.032. — PMID 17081978.
- Yoshikawa M., Peragine A., Park M. Y., Poethig R. S. A pathway for the biogenesis of trans-acting siRNAs in Arabidopsis. (англ.) // Genes & development. — 2005. — Vol. 19, no. 18. — P. 2164—2175. — doi:10.1101/gad.1352605. — PMID 16131612.
- Talmor-Neiman M., Stav R., Klipcan L., Buxdorf K., Baulcombe D. C., Arazi T. Identification of trans-acting siRNAs in moss and an RNA-dependent RNA polymerase required for their biogenesis. (англ.) // The Plant journal : for cell and molecular biology. — 2006. — Vol. 48, no. 4. — P. 511—521. — doi:10.1111/j.1365-313X.2006.02895.x. — PMID 17076803.
- Xie Z., Qi X. Diverse small RNA-directed silencing pathways in plants. (англ.) // Biochimica et biophysica acta. — 2008. — Vol. 1779, no. 11. — P. 720—724. — doi:10.1016/j.bbagrm.2008.02.009. — PMID 18358247.
- Allen E., Xie Z., Gustafson A. M., Carrington J. C. microRNA-directed phasing during trans-acting siRNA biogenesis in plants. (англ.) // Cell. — 2005. — Vol. 121, no. 2. — P. 207—221. — doi:10.1016/j.cell.2005.04.004. — PMID 15851028.
- Allen E., Howell M. D. miRNAs in the biogenesis of trans-acting siRNAs in higher plants. (англ.) // Seminars in cell & developmental biology. — 2010. — Vol. 21, no. 8. — P. 798—804. — doi:10.1016/j.semcdb.2010.03.008. — PMID 20359543.
- Tomari Y., Zamore P. D. Perspective: machines for RNAi. (англ.) // Genes & development. — 2005. — Vol. 19, no. 5. — P. 517—529. — doi:10.1101/gad.1284105. — PMID 15741316.
- Adenot X., Elmayan T., Lauressergues D., Boutet S., Bouché N., Gasciolli V., Vaucheret H. DRB4-dependent TAS3 trans-acting siRNAs control leaf morphology through AGO7. (англ.) // Current biology : CB. — 2006. — Vol. 16, no. 9. — P. 927—932. — doi:10.1016/j.cub.2006.03.035. — PMID 16682354.
- Wu L., Mao L., Qi Y. Roles of dicer-like and argonaute proteins in TAS-derived small interfering RNA-triggered DNA methylation. (англ.) // Plant physiology. — 2012. — Vol. 160, no. 2. — P. 990—999. — doi:10.1104/pp.112.200279. — PMID 22846193.
- Williams L., Carles C. C., Osmont K. S., Fletcher J. C. A database analysis method identifies an endogenous trans-acting short-interfering RNA that targets the Arabidopsis ARF2, ARF3, and ARF4 genes. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2005. — Vol. 102, no. 27. — P. 9703—9708. — doi:10.1073/pnas.0504029102. — PMID 15980147.
- Heisel S. E., Zhang Y., Allen E., Guo L., Reynolds T. L., Yang X., Kovalic D., Roberts J. K. Characterization of unique small RNA populations from rice grain. (англ.) // Public Library of Science ONE. — 2008. — Vol. 3, no. 8. — P. e2871. — doi:10.1371/journal.pone.0002871. — PMID 18716673.
Литература
- Zhang C., Li G., Zhu S., Zhang S., Fang J. tasiRNAdb: a database of ta-siRNA regulatory pathways. (англ.) // Bioinformatics. — 2014. — Vol. 30, no. 7. — P. 1045—1046. — doi:10.1093/bioinformatics/btt746. — PMID 24371150.
- Li F., Orban R., Baker B. SoMART: a web server for plant miRNA, tasiRNA and target gene analysis. (англ.) // The Plant journal : for cell and molecular biology. — 2012. — Vol. 70, no. 5. — P. 891—901. — doi:10.1111/j.1365-313X.2012.04922.x. — PMID 22268718.
- Matsui A., Mizunashi K., Tanaka M., Kaminuma E., Nguyen A. H., Nakajima M., Kim J. M., Nguyen D. V., Toyoda T., Seki M. tasiRNA-ARF pathway moderates floral architecture in arabidopsis plants subjected to drought stress. (англ.) // BioMed research international. — 2014. — Vol. 2014. — P. 303451. — doi:10.1155/2014/303451. — PMID 25243128.
- Fahlgren N., Montgomery T. A., Howell M. D., Allen E., Dvorak S. K., Alexander A. L., Carrington J. C. Regulation of AUXIN RESPONSE FACTOR3 by TAS3 ta-siRNA affects developmental timing and patterning in Arabidopsis. (англ.) // Current biology : CB. — 2006. — Vol. 16, no. 9. — P. 939—944. — doi:10.1016/j.cub.2006.03.065. — PMID 16682356.
