ПЭТаза
ПЭТазы представляют собой класс ферментов эстераз, которые катализируют гидролиз полиэтилентерефталатного (ПЭТ) пластика до мономерного моно-2-гидроксиэтилтерефталата (MHET).
| ПЭТаза | |
|---|---|
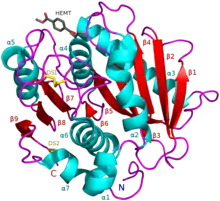 ПЭТаза I. sakaiensis в комплексе с HEMT, аналогом ПЭТ (PDB 5XH3). | |
| Идентификаторы | |
| Шифр КФ | 3.1.1.101 |
| Базы ферментов | |
| IntEnz | IntEnz view |
| BRENDA | BRENDA entry |
| ExPASy | NiceZyme view |
| MetaCyc | metabolic pathway |
| KEGG | KEGG entry |
| PRIAM | profile |
| PDB structures | RCSB PDB PDBe PDBj PDBsum |
| Поиск | |
| PMC | статьи |
| PubMed | статьи |
| NCBI | NCBI proteins |
Идеализированная химическая реакция (где n — количество мономеров в полимерной цепи):[1]
- (этилентерефталат)n + H2O → (этилентерефталат)n-1 + MHET
Остаточный ПЭТ распадается на бис (2-гидроксиэтил) терефталат (BHET). ПЭТазы также могут разрушать ПЭФ-пластик (полиэтилен-2,5-фурандикарбоксилат), который является биоразлагаемой заменой ПЭТ. ПЭТазы не могут катализировать гидролиз алифатических полиэфиров, таких как полибутиленсукцинат или полимолочная кислота[2].
Неферментативное естественное разложение ПЭТ займёт сотни лет, но ПЭТазы могут разложить мелкодисперсный ПЭТ-пластик за считанные дни[3].
История
Первая ПЭТаза была обнаружена в 2016 году в бактериях штамма 201-F6 Ideonella sakaiensis, обнаруженных в образцах осадка, собранных недалеко от японского предприятия по переработке ПЭТ-бутылок[1][4]. До этого открытия были известны другие типы гидролаз, разлагающих ПЭТ[2]. К ним относятся такие гидролазы, как липазы, эстеразы и кутиназы[5]. Открытия ферментов, разлагающих полиэфир, датируются, по крайней мере, 1975 г. (α-химотрипсин)[6] и 1977 г. (липаза), например[7].
ПЭТ-пластик получил широкое распространение в 1970-х годах, и было высказано предположение, что ПЭТазы в бактериях появились только недавно[2]. В прошлом у ПЭТазы могла быть ферментативная активность, связанная с разрушением воскового покрытия растений[8].
Структура
По состоянию на апрель 2019 года было известно 17 трёхмерных кристаллических структур ПЭТаз: 6QGC, 6ILX, 6ILW, 5YFE, 6EQD, 6EQE, 6EQF, 6EQG, 6EQH, 6ANE, 5XJH, 5YNS, 5XFY, 5XFZ, 5XG0, 5XH2 и 5XH2. .
ПЭТаза обладает общими качествами, как с липазами, так и с кутиназами в том, что она обладает α/β-гидролазной складкой; тем не менее, расщелина активного центра, наблюдаемая в ПЭТазе, более открыта, чем в кутиназах[2]. Согласно Pfam, ПЭТаза Ideonella sakaiensis похожа на диенелактонгидролазу. Согласно ESTHER, он относится к семейству полиэстераза-липаза-кутиназа.
Существует приблизительно 69 ферментов, подобных ПЭТазе, найденных у множества различных организмов, и существует две классификации этих ферментов, включая тип I и тип II. Предполагается, что 57 ферментов относятся к категории I типа, тогда как остальные относятся к группе II типа, включая фермент ПЭТазы, обнаруженного у Ideonella sakaiensis. Во всех 69 ферментах, подобных ПЭТазе, существуют одни и те же три остатка в активном центре, что позволяет предположить, что каталитический механизм одинаков для всех форм ферментов, подобных ПЭТазе[9].
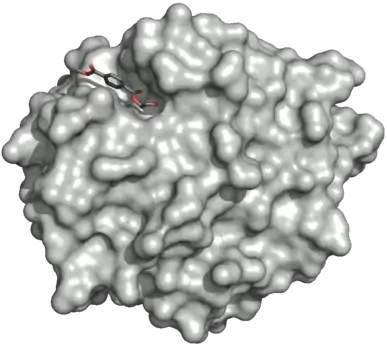 Поверхность двойного мутанта петазы (R103G и S131A) с HEMT (1-(2-гидроксиэтил) 4-метилтерефталатом), связанного с его активным сайтом. HEMT является аналогом MHET и содержит дополнительный этерифицированный метанол. PDBID: 5XH3.
Поверхность двойного мутанта петазы (R103G и S131A) с HEMT (1-(2-гидроксиэтил) 4-метилтерефталатом), связанного с его активным сайтом. HEMT является аналогом MHET и содержит дополнительный этерифицированный метанол. PDBID: 5XH3.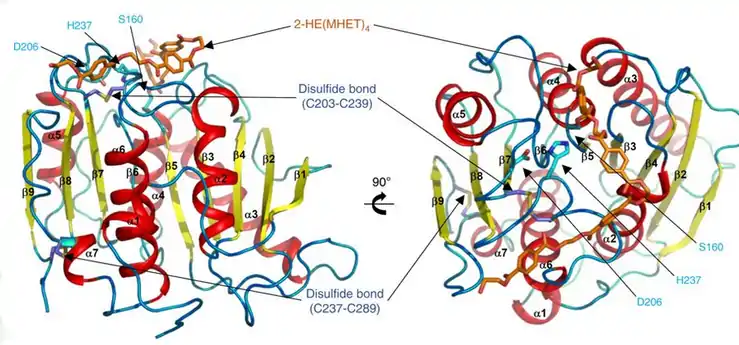 Ленточная диаграмма ПЭТазы с тремя остатками Ser160, Asp206 и His237. Каталитическая триада представлена палочками голубого цвета. Активный сайт показан оранжевым цветом, для представления стимуляции молекулой 2-HE(MHET)4[9].
Ленточная диаграмма ПЭТазы с тремя остатками Ser160, Asp206 и His237. Каталитическая триада представлена палочками голубого цвета. Активный сайт показан оранжевым цветом, для представления стимуляции молекулой 2-HE(MHET)4[9].
Мутации
В 2018 году учёные из Портсмутского университета в сотрудничестве с Национальной лабораторией возобновляемых источников энергии Министерства энергетики США разработали мутант ПЭТазы, который разлагает ПЭТ быстрее, чем тот, который находится в естественном состоянии. В этом исследовании также было показано, что ПЭТазы могут разрушать полиэтилен-2,5-фурандикарбоксилат (ПЭФ)[2][10].
Биологический путь

У I. sakaiensis образующийся MHET дополнительно расщепляется под действием фермента MHETаза на терефталевую кислоту и этиленгликоль[1]. Лабораторные эксперименты показали, что химерные белки, которые искусственно связывают MHETазу и ПЭТазу, превосходят аналогичные смеси свободных ферментов[12].
См. также
- Galleria mellonella , гусеница, способная переваривать полиэтилен .
- Aspergillus tubingensis — гриб, способный переваривать полиуретан .
- Pestalotiopsis microspora, вид эндофитного гриба, способный расщеплять полиуретан.
- Кутиназа, фермент эстераза аналогичной геометрической формы
Примечания
- “A bacterium that degrades and assimilates poly(ethylene terephthalate)”. Science. 351 (6278): 1196—9. March 2016. DOI:10.1126/science.aad6359. PMID 26965627.
- “Characterization and engineering of a plastic-degrading aromatic polyesterase”. Proceedings of the National Academy of Sciences of the United States of America. 115 (19): E4350–E4357. May 2018. DOI:10.1073/pnas.1718804115. PMID 29666242.
- Dockrill. Scientists Have Accidentally Created a Mutant Enzyme That Eats Plastic Waste (англ.), ScienceAlert. Дата обращения 27 ноября 2018.
- “Ideonella sakaiensis sp. nov., isolated from a microbial consortium that degrades poly(ethylene terephthalate)”. International Journal of Systematic and Evolutionary Microbiology. 66 (8): 2813—8. August 2016. DOI:10.1099/ijsem.0.001058. PMID 27045688.
- “Structural insight into catalytic mechanism of PET hydrolase”. Nature Communications []. 8 (1): 2106. December 2017. DOI:10.1038/s41467-017-02255-z. PMID 29235460.
- Tabushi, Iwao (August 1975). “Polyester readily hydrolyzable by chymotrypsin”. Journal of Polymer Science: Polymer Letters Edition. 13 (8): 447—450. DOI:10.1002/pol.1975.130130801.
- “Hydrolysis of polyesters by lipases”. Nature. 270 (5632): 76—8. November 1977. DOI:10.1038/270076a0. PMID 927523.
- Lab 'Accident' Becomes Mutant Enzyme That Devours Plastic, Live Science. Дата обращения 27 ноября 2018.
- “Structural insight into molecular mechanism of poly(ethylene terephthalate) degradation”. Nature Communications []. 9 (1): 382. January 2018. DOI:10.1038/s41467-018-02881-1. PMID 29374183.
- Carrington. New super-enzyme eats plastic bottles six times faster (англ.), The Guardian.
- Allison Chan. The Future of Bacteria Cleaning Our Plastic Waste (2016).
- “Characterization and engineering of a two-enzyme system for plastics depolymerization”. Proc Natl Acad Sci U S A. 117 (41): 25476—25485. Oct 2020. DOI:10.1073/pnas.2006753117. PMID 32989159.
