Открытый хроматин
Откры́тый хромати́н (англ. open chromatin) — небольшие участки хроматина, свободные от нуклеосом[1]. Посадке нуклеосом, как правило, препятствуют связанные с хроматином белковые факторы, узнающие определённые последовательности ДНК. К числу таких белков относятся транскрипционные факторы, ДНК- или РНК-полимеразы. Открытый хроматин часто совпадает с цис-регуляторными последовательностями, а именно: промоторами, энхансерами, инсуляторами, сайленсерами, участками начала репликации ДНК[2]. Размер открытых участков хроматина обычно составляет несколько сотен пар нуклеотидов, в среднем около 300 п.н[3].
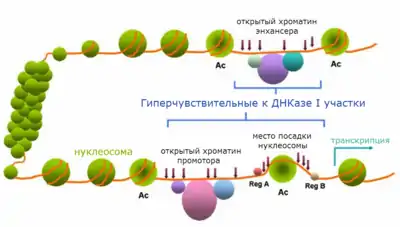
Открытый хроматин наиболее часто определяют с помощью метода ДНКазной чувствительности. Свободные от нуклеосом участки хроматина предпочтительно атакуются ДНКазой I при обработке ей пермеабилизованных клеток или изолированных ядер. В связи с этим открытый хроматин часто называют гиперчувствительными к ДНКазе I участками, или гиперчувствительными сайтами (англ. hypersensitive sites). Вероятность расщепления ДНК нуклеазой в гиперчувствительных сайтах может превосходить среднестатистическую в сотни и даже тысячи раз. Гиперчувствительность к ДНКазе I открытого хроматина следует отличать от повышенной общей ДНКазной чувствительности активно транскрибирующихся генов[4].
В зависимости от типа белковых факторов, связывание которых с ДНК препятствует посадке нуклеосом, гиперчувствительные к ДНКазе I участки хроматина могут быть тканеспецифичными или конститутивными, то есть присутствующими в клетках, дифференцированных по разным путям.
Картирование областей открытого хроматина
Для картирования областей открытого хроматина используют методы ДНКазной чувствительности (англ. DNase I hypersensitive) и изоляции регуляторных элементов с помощью формальдегида англ. formaldehyde-assisted isolation of regulatory elements (FAIRE)[1]. Метод ДНКазной чувствительности не позволяет определить, каким именно регуляторным участком является данная область открытого хроматина[1].
Ранее анализ результатов метода ДНКазной чувствительности проводился с помощью саузерн-блот гибридизации (англ. Southern blot). Это не позволяло проводить анализ большого количества сайтов, а также находить новые сайты гиперчувствительности. Анализ ДНКазной чувствительности можно проводить также с помощью ПЦР в реальном времени (количественной ПЦР). Это значительно проще, чем саузерн-блот гибридизация, но этот метод также имеет ограничение по количеству сайтов для анализа и не может быть использован для полногеномного исследования распределения сайтов чувствительности к ДНКазе I[5].
Развитие методов высокоэффективного секвенирования (англ. High-throughput sequencing) и ДНК-микрочипов (англ. DNA-microarray) позволяет картировать области открытого хроматина на всем протяжении генома[6]. Кроме того, сочетание метода ДНКазной чувствительности с методом иммунопреципитации хроматина (англ. Chromatin immunoprecipitation (ChIP)) с последующим высокоэффективным секвенированием позволяет получать больше информации о связывании конкретных транскрипционных факторов с активными участками хроматина[1].
Другой способ картирования областей открытого хроматина — проведение иммунопреципитации хроматина (англ. ChIP) на антитела к гистонам. При этом области открытого хроматина должны быть мало представлены, так как с ними не связаны нуклеосомы. Метод ДНКазной чувствительности и иммунопреципитация гистонов дают сходные результаты[7].
Значение открытого хроматина
В геномах эукариот некодирующие последовательности, участвующие в регуляции экспрессии генов на разных стадиях развития организма или в разных тканях, приобретают особое значение. Открытие и характеристика регуляторных участков становится необходимой для понимания закономерностей в экспрессии генов[5]. Так в геноме человека более 95 % ДНК является некодирующей. В этот класс последовательностей, кроме мусорной ДНК, входят важные регуляторные последовательности: промоторы, энхансеры, сайленсеры, инсуляторы или локусы контроля (англ. locus control regions (LCR)). ENCODE консорциум показал, что сайты гиперчувствительности к ДНКазе I, идентифицированные в 1 % генома человека, являются маркерами модификаций гистонов, участков ранней репликации, сайтов начала транскрипции и сайтами связывания транскрипционных факторов[8]. Также открытый хроматин часто связан с активно транскрибируемыми некодирующими РНК[8].
Распределение открытого хроматина
Кроме некодирующих регуляторных последовательностей, открытый хроматин также ассоциирован с экзонами и интронами активно транскрибируемых генов. Особенно часто такие участки открытого хроматина совпадают первым экзоном и интроном гена[5]. Однако наличие открытого хроматина не является достаточным условием активности гена. Нетранскрибируемые гены, связанные с открытым хроматином, находятся в состоянии «готовности» к транскрипции (англ. poised state)[5]. Таким образом, формирование открытого хроматина или перевод в неактивное состояние является важным для регуляции экспрессии генов.
Свободными от нуклеосом могут быть не только участки связывания транскрипционных факторов и других регуляторных белков. Некоторые последовательности ДНК не способны наматываться на нуклеосомные глобулы. Это последовательности, обладающие пониженной гибкостью, и последовательности, склонные с созданию неканонических структур, например, шпилек[9].
Из рисунка, представляющего скриншот геномного браузера UCSC, видна колокализация сайта ДНКазной гиперчувствительности (англ. DNaseI Hypersensitivity Clusters) с промоторами двух генов. Области открытого хроматина окружены гистонами H3, ацетилироваными по 27-му остатку лизина (H3K27Ac), что является меткой активных регуляторных областей хроматина, таких как промоторы и энхансеры. Кроме того, в районе сайта ДНКазной гиперчувствительности находится сайт связывания многих транскрипционных факторов, среди которых можно обнаружить консервативный фактор инициации транскрипции TBP (является основной частью TFIID). Также можно заметить частое связывание в этом районе РНК-полимеразы II, осуществляющей транскрипцию белок-кодирующих генов у человека. Для данного сайта ДНКазной гиперчувствительности характерна повышенная консервативность среди млекопитающих (англ. Mammal Cons), что означает сохранение этой последовательности в ходе эволюции, и, как следствие, её функциональное значение[8].
Ремоделирование хроматина
Образование областей, свободных от нуклеосом, происходит под действием специальных факторов, осуществляющих сборку, разборку и перемещение нуклеосом. Процесс изменения положения нуклеосом называется ремоделированием хроматина. В нём участвуют комплексы ремоделирования хроматина — консервативные белковые комплексы, работающие с затратой энергии АТФ. Ремоделирование хроматина осуществляется после внесения определенных эпигенетических меток — модификации гистонов или метилирования ДНК. Если метки соответствуют активному хроматину (например, ацетилированый 9-й лизин гистона H3, ди- и триметилированный 4-й лизин гистона H3 и многие другие), то образуются участки открытого хроматина. Часто профиль модификаций гистонов имеет определенное распределение вокруг сайта гиперчувствительности к ДНКазе I[5].
Открытый хроматин в различных тканях и клетках
Высокоэффективные методы позволяют сравнивать распределение областей открытого хроматина в различных тканях или культурах клеток из одного организма. Подобное сравнение выявляет существенное различие в распределении таких участков в геноме[5]. Это говорит о различной активности таких участков в разных тканях. Так промотор и энхансер гена могут находиться в области открытого хроматина в одной ткани и быть закрыты нуклеосомами в другой. Это говорит о различной экспрессии генов в разных тканях и наиболее характерно для тканеспецифичных (англ. tissue-specific) генов. Наоборот, гены, находящиеся в области открытого хроматина во всех тканях и клеточных линиях, обычно, относятся к генам домашнего хозяйства (англ. housekeeping genes). Также может происходить изменение профиля ДНКазной чувствительности в процессе развития и дифференцировки клеток. Для выявления активности тканеспецифичных генов используют определение генной онтологии (англ. Gene Ontology (GO)) после проведения DNase-seq[5].
Примечания
- Boyle A. P., Furey T. S. High-resolution mapping studies of chromatin and gene regulatory elements. (англ.) // Epigenomics. — 2009. — Vol. 1, no. 2. — P. 319—329. — doi:10.2217/epi.09.29. — PMID 20514362.
- Разин, 2009, с. 21—24.
- ENCODE concortium. The downloadable files associated with the ENCODE Regulation 'DNase Clusters' track (англ.). ENCODE concortium. Дата обращения: 25 апреля 2013. Архивировано 30 апреля 2013 года.
- Разин, 2009, с. 43.
- Boyle A. P., Davis S., Shulha H. P., Meltzer P., Margulies E. H., Weng Z., Furey T. S., Crawford G. E. High-resolution mapping and characterization of open chromatin across the genome. (англ.) // Cell. — 2008. — Vol. 132, no. 2. — P. 311—322. — doi:10.1016/j.cell.2007.12.014. — PMID 18243105.
- Lee K., Kim S. C., Jung I., Kim K., Seo J., Lee H. S., Bogu G. K., Kim D., Lee S., Lee B., Choi J. K. Genetic landscape of open chromatin in yeast. (англ.) // PLoS genetics. — 2013. — Vol. 9, no. 2. — P. e1003229. — doi:10.1371/journal.pgen.1003229. — PMID 23408895.
- Bartkuhn M., Straub T., Herold M., Herrmann M., Rathke C., Saumweber H., Gilfillan G. D., Becker P. B., Renkawitz R. Active promoters and insulators are marked by the centrosomal protein 190. (англ.) // The EMBO journal. — 2009. — Vol. 28, no. 7. — P. 877—888. — doi:10.1038/emboj.2009.34. — PMID 19229299.
- Birney E., Stamatoyannopoulos J. A., Dutta A. et al. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project. (англ.) // Nature. — 2007. — Vol. 447, no. 7146. — P. 799—816. — doi:10.1038/nature05874. — PMID 17571346.
- Разин, 2009, с. 23.
Литература
- Разин С. В. Хроматин: упакованный геном / С. В. Разин, А. А. Быстрицкий. — М.: БИНОМ: Лаборатория знаний, 2009. — 176 с. — ISBN 978-5-9963-0087-7.