ДНК-микрочип
ДНК-микрочип, или ДНК-чип (англ. DNA microarray) — технология, используемая в молекулярной биологии и медицине. ДНК-микрочип представляет собой множество небольших одноцепочечных молекул — ДНК-зондов, которые ковалентно пришиты к твёрдому основанию[1][2]. Каждый такой зонд имеет строго определённую последовательность нуклеотидов и место на микрочипе. Одинаковые зонды располагаются вместе, образуя сайт микрочипа. Между сайтом и последовательностью ДНК зонда есть взаимно-однозначное соответствие. ДНК-микрочипы используются для определения ДНК или РНК (обычно после обратной транскрипции), которые могут быть как белок-кодирующими, так и не кодировать белки. Измерение генной экспрессии посредством кДНК называется профилем экспрессии, или экспрессионным анализом. На современных микрочипах можно полностью расположить целый геном, каждый известный ген которого будет являться зондом[3].

Принцип метода
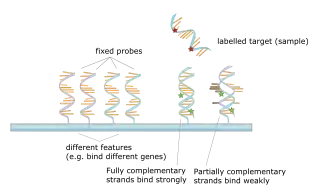
В основе работы ДНК-микрочипов лежит явление гибридизации. При наличии небольших количеств ДНК исследуемого образца осуществляют амплификацию. Для РНК сначала осуществляется обратная транскрипция, что впрочем необязательно: существуют чипы, работающие как с ДНК, так и с РНК. Проверка образцов ДНК/РНК заключается в мечении образцов различными флуоресцентными метками для последующего обнаружения и нанесении образцов на микрочип. ДНК-микрочип с нанесённым на него образцом инкубируют некоторое время, чтобы произошла гибридизация комплементарных одноцепочечных молекул, после чего чип промывают. Все некомплементарные ДНК/РНК образца смываются с чипа. После этого производят сканирование микрочипа при помощи лазера, который вызывает флуоресценцию меченных молекул образца. Подключённый к компьютеру микроскоп оценивает флуоресценцию каждого сайта ДНК-микрочипа, а следовательно и устанавливает последовательности гибридизованных ДНК, что позволяет установить последовательность ДНК,РНК из образца[4].
История
Технология ДНК-микрочипов берёт начало от Саузерн-блоттинга — методики, в которой фрагментированную ДНК переносят на подходящий носитель и затем с помощью зонда с известной нуклеотидной последовательностью определяют содержание целевой последовательности в образце. Впервые набор различных ДНК, объединённых в чип, был использован в 1987 году для определения особенностей регуляции экспрессии генов интерферонами[5]. Ранние ДНК-микрочипы были сделаны путём «раскапывания» микроколичеств кДНК на фильтровальную бумагу. Использование миниатюрных чипов для определения особенностей экспрессии генов было осуществлено в 1995 году[6] и полный эукариотический геном (Saccharomyces cerevisiae) был размещён на микрочипе в 1997 году[3].
Сборка ДНК-микрочипов
Амплифицированные фрагменты ДНК при помощи микроманипулятора наносят на кремниевые или стеклянные пластины, ковалентно закрепляя зонды. В 1991—1993 годах был предложен другой подход, в основе которого лежала технология фотолитографии, используемая в полупроводниковой индустрии. Позже, в 1997 году, был запатентован ещё один метод — «электрофокусирование»[2].
Фотолитография
Сборка начинается с нанесения на стеклянную пластинку размером 12,8×12,8 мм специального защитного фоточувствительного слоя, который делает саму пластинку инертной. Только те места, где защитный слой будет освещён, способны присоединить дезоксирибонуклеотиды (A, T, G, C). После воздействия света на пластинку наносится раствор одного из оснований. Все остальные места защищены специальной фотолитографической маской, таким образом, дезоксирубонуклеотиды ковалентно присоединяются к пластинке в точно нужных местах, после чего все, что не смогло связаться — смывается. Сами нуклеотиды химически модифицированы так, чтобы они могли связать другой нуклеотид только при воздействии света[7]. Многократно повторяя циклы маски — освещения — нанесение нуклеотида — промывания столько раз сколько необходимо и постоянно меняя маски можно добиться создания уникальных последовательностей. Различные маски разрабатывают для создания чипа, в соответствии с требованиями к получаемым зондам[2].
Цепи одинаковых одноцепочных ДНК, организованные в квадрат 90х90 мкм, растут на поверхности пластины нуклеотид за нуклеотидом. Каждый квадрат в итоге содержит миллиарды идентичных зондов. Сейчас фотолитографию используют для создания относительно коротких зондов, длиной не более 100 нуклеотидов. При этом количество масок, сменяемых за время сборки такого чипа сопоставимо с длиной зондов: так, для сборки зондов длиной 18-25 нуклеотидов на чипе потребуется порядка 40 масок. На практике большое число идентичных ДНК-микрочипов изготавливаются вместе на большой стеклянной подложке[2].
Электрофокусирование
Сборка такого рода микрочипов требует особенно сложных подложек, представляющих собой по сути электронные чипы, со множеством выходов, каждый из которых контролирует напряжение на определённом месте пластинки. В отличие от фотолитографии, где зонды собираются основание за основанием, здесь уже готовые одноцепочечные олигонуклеотиды доставляются к нужным местам пластинки под действием электрического поля. В нужном месте пластинки при помощи соответствующего выхода микрочипа создается положительное напряжение. Отрицательно заряженная одноцепочечная ДНК движется к созданному положительному заряду и прикрепляется в нужном месте. После этого активируется следующий выход микрочипа, давая положительный заряд в другом месте пластинки, куда и направляется следующий зонд. Плотность расположения ДНК на такого рода рода чипах гораздо меньше чем на чипах, полученных при помощи фотолитографии[8][2].
Эксперимент
Существует три базовых типа микрочипов: для анализа генной экспрессии (GEM-microarray), для сравнительной геномной гибридизации (MCGH) и для обнаружения единичных нуклеотидных полиморфизмов (SNPM)[9]. Базовый протокол может быть представлен в следующем виде[10]:
- 1. Выделение ДНК/РНК
На этом этапе выделяется либо мРНК (GEM), либо фрагменты геномной ДНК (MCGH, SNPM) из интересующих образцов. Сейчас это легко осуществляется специальными коммерческими наборами[10][11].
- 2. Мечение ДНК/РНК
Этот процесс начинается с обратной транскрипции (если необходимо). Затем производят амплификацию целевого фрагмента при помощи полимеразной цепной реакции. В процессе ПЦР в состав фрагментов ДНК включаются нуклеотиды с флуоресцентными метками[10][12].
- 3. Гибридизация
Здесь флуоресцентно-меченные амплифицированные образцы используются как мишени, для поиска на микрочипе комплементарных цепей, то есть способных образовывать прочные двойные цепи — дуплексы, по правилу комплементарности. Как пример, комплементарные последовательности 5'-GCATGCAT-3’ и 3’-CGTACGTA-5’. Так как одна из цепей образуемого дуплекса меченая, то сигнал от такого дуплекса можно зарегистрировать[10].
- 4. Промывка
Как только заканчивается гибридизация, чип вынимают из раствора, содержащего образцы меченой ДНК. Сам чип потом тщательно промывается по определённым методикам, используя различные буферные смеси, центрифугирование и так далее[10].
- 5. Сканирование
Этот процесс предполагает использование оптических сканирующих проборов, способных детектировать фотоны с точно определённой длинной волны. Каждый сайт чипа освещается пучком света определённой длины волны, что активирует флуоресцентую метку. «Просвечивание» микрочипа производят аргоновыми лазерами. Активированная флуоресцентная метка испускает фотон чуть большей длины волны, который регистрирует прибор. Чем больше фотонов при одном засвете уловит прибор, тем выше интенсивность свечения данной точки, а значит там большое число образовавшихся дуплексов[10].
- 6. Анализ данных
Данные — массив значений интенсивности свечения каждого конкретного сайта микрочипа. Используя математические методы сравнения, можно достоверно установить сайты чипа, где произошла гибридизация, а значит и узнать последовательность ДНК/РНК из образца[10][13].
Области применения
Анализ экспрессии генов
Это самое частое применение ДНК-микрочипов. РНК, выделенная из культуры клеток, подвергается обратной транскрипции, в результате которой получается меченная кДНК. Иногда требуется ещё один этап транскрипции с кДНК (для чипов, работающих с РНК), при котором создается меченная кРНК. Существует несколько различных способов внести метки в целевую молекулу: включение флуоресцентно меченных нуклеотидов в процессе синтеза кДНК или кРНК, использование биотин-модифицированных нуклеотидов, которые потом окрашиваются флуоресцентно-меченным стрептавидином, использование во время синтеза модифицированных нуклеотидов, на которые затем можно добавить флуоресцентную метку[14].
Меченная таким образом ДНК или РНК гибридизуется на микрочипе, после чего смывается. В каждой точке чипа детектируется флуоресцентный сигнал. В случае с биотинилированными образцами ДНК-микрочип после гибридизации окрашивается стрептавидином, содержащим флуоресцентые метки. Флуоресценция возбуждается светом лазера и регистрируется, как правило, сканирующим конфокальном микроскопом[15].
Анализ связывания транскрипционных факторов
Микрочипы также используются вместе с иммунопреципитацией хроматина для определения сайтов связывания транскрипционных факторов(ТФ)[16][17]. В экстракт клеточной ДНК добавляют формальдегид, что ведет к образованию ковалентных сшивок между ДНК и белками. После чего ДНК фрагментируется. Нужный ТФ выделяется из смеси при помощи аффинной хроматографии с использованием антител или тэгов, которые вставляются в данный ТФ генно-инженерными методами заранее. После очистки ДНК освобождается от ТФ, амплифицируется, флуоресцентно метится и используется для гибридизации на микрочипе. Такая техника широко известна как «ChIP-chip»[18], или иммунопреципитация хроматина на микрочипе, но у нее есть ограничения в связи с тем, что ТФ могут связываться далеко от регулируемого ими гена.
Генотипирование
ДНК-микрочипы широко используются для выявления однонуклеотидных полиморфизмов (SNP). Существует несколько различных подходов[15]:
Аллельная дискриминация
На микрочипах располагаются короткие зонды (25 нуклеотидов для микрочипов Affymetrix), содержащие все варианты SNP в центре, так как эта позиция наиболее сильно влияет на качество гибридизации. Фрагментированная, амплифицированная, флуоресцентно-меченная ДНК образца наносится на микрочип, где происходит гибридизация образца и зонда. В местах полной комплементарности молекул регистрируется сильный сигнал[19].
«Golden Gate» анализ
В основе этого метода лежит полимеразная цепная реакция. В раствор геномной ДНК помещаются молекулы комплементарные геномной ДНК и на 3'-конце содержащие различные модификации SNP, а на 5'-конце различные праймеры для последующего проведения ПЦР, кроме того добавляется и комплементарная другой цепи геномной ДНК молекула с другой стороны от SNP, содержащая на 5'-конце ещё один праймер для ПЦР. Полимераза будет осуществлять синтез только с того праймера, 3'-конец которого соответствует SNP. В итоге в зависимости от того, с какого праймера с видоизмененным 3'-концом произойдёт ПЦР, такой SNP и наблюдается в образце[20].
Расширение праймеров
Здесь зонды подобраны так, что покрывают весь участок ДНК прямо до SNP, не включая полиморфизм. Фрагментированная геномная ДНК гибридизуется с таким чипом, после чего в раствор добавляется полимераза и флуоресцентно-меченные нуклеотиды с 4 различными метками на 3'-конце. Подобные нуклеотиды могут присоединиться полимеразой к имеющейся цепи, но потом присоединить к себе следующий нуклеотид не могут. В итоге зонды расширяются на один нуклеотид, который и соответствует SNP[21].
Оценка крайнего нуклеотида
Этот метод схож с методом расширения праймеров, с тем отличием, что зонды располагаются не на плоской подложке, а на множестве мелких шариков. SNP также распознается по цвету метки единственного достроенного нуклеотида[22].
Ограничения
Несмотря на достоинства ДНК-микрочипов, у них есть и ограничения в применении. Предполагается, что интенсивность сигнала (свечения), зарегистрированная в конкретном сайте микрочипа, линейно зависит от количества ДНК, которое прошло гибридизацию, что не всегда так: из-за кинетики гибридизации уровень сигнала, полученный в данной точке, не является линейной функцией от концентрации данной ДНК в образце. Таким образом, точно оценивать количество ДНК в образце можно лишь в определённом диапазоне изначальных концентраций ДНК, которые всё же могут обеспечить линейную зависимость. Оценка относительно изначально больших или малых концентраций ДНК образца будет неточной[15].
В сложных геномах эукариот, особенно млекопитающих, встречается множество гомологичных генов, последовательности которых очень сходны, что накладывает дополнительные условия на дизайн зондов для микрочипов[23][24]. Зонд, сконструированный для одного гена A, может также «поймать на себя» ещё и гены B, C, D, оказавшиеся гомологичными гену A, что исказит итоговую картину.
Ещё одно ограничение связано с объемом базы известных генов. Невозможен синтез зонда, соответствующего неизвестному гену, и никакие взаимодействия таких генов не могут быть обнаружены. Эта проблема особенно актуальна для прокариот, так как геномы даже близкородственных организмов могут значительно отличаться. Например, у вида бактерий Aggregatibacter actinomycetemcomitans геномы различных штаммов могут различаться на 20% генов и, следовательно, микрочипы, сконструированные под один штамм, не смогут обнаружить другой[25].
См. также
Примечания
- Javier Garaizar, Aitor Rementeria, Steffen Porwollik. DNA microarray technology: a new tool for the epidemiological typing of bacterial pathogens? // FEMS immunology and medical microbiology. — 2006-07-01. — Т. 47, вып. 2. — С. 178—189. — ISSN 0928-8244. — doi:10.1111/j.1574-695X.2006.00081.x.
- M. Bednár. DNA microarray technology and application // Medical Science Monitor: International Medical Journal of Experimental and Clinical Research. — 2000-07-01. — Т. 6, вып. 4. — С. 796—800. — ISSN 1234-1010.
- Lashkari D.A., DeRisi J.L., McCusker J.H., Namath A.F., Gentile C., Hwang S.Y., Brown P.O., Davis R.W. Yeast microarrays for genome wide parallel genetic and gene expression analysis (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1997. — Vol. 94. — P. 13057—13062. — doi:10.1073/pnas.94.24.13057. — PMID 9371799.
- Б. Альбертс. Молекулярная биология клетки: в 3 томах.. — 2013. — С. 881-885. — 2821 с. — ISBN 978-0-8153-4111-6. — ISBN 978-5-4344-0137-1.
- Kulesh D.A., Clive D.R., Zarlenga D.S., Greene J.J. Identification of interferon-modulated proliferation-related cDNA sequences (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1987. — Vol. 84. — P. 8453—8457. — doi:10.1073/pnas.84.23.8453. — PMID 2446323.
- Schena M., Shalon D., Davis R.W., Brown P.O. Quantitative monitoring of gene expression patterns with a complementary DNA microarray (англ.) // Science : journal. — 1995. — Vol. 270. — P. 467—470. — doi:10.1126/science.270.5235.467. — PMID 7569999.
- G. McGall, J. Labadie, P. Brock, G. Wallraff, T. Nguyen. Light-directed synthesis of high-density oligonucleotide arrays using semiconductor photoresists (англ.) // Proceedings of the National Academy of Sciences. — United States National Academy of Sciences, 1996-11-26. — Vol. 93, iss. 24. — P. 13555—13560. — ISSN 0027-8424.
- C. F. Edman, D. E. Raymond, D. J. Wu, E. Tu, R. G. Sosnowski. Electric field directed nucleic acid hybridization on microchips // Nucleic Acids Research. — 1997-12-15. — Т. 25, вып. 24. — С. 4907—4914. — ISSN 0305-1048.
- National Center for Biotechnology Information. MICROARRAYS: CHIPPING AWAY AT THE MYSTERIES OF SCIENCE AND MEDICINE.
- Dan Tulpan. Recent patents and challenges on DNA microarray probe design technologies // Recent patents on DNA & gene sequences. — 2010-11-01. — Т. 4, вып. 3. — С. 210—217. — ISSN 2212-3431.
- Siun Chee Tan, Beow Chin Yiap. DNA, RNA, and protein extraction: the past and the present // Journal of Biomedicine & Biotechnology. — 2009-01-01. — Т. 2009. — С. 574398. — ISSN 1110-7251. — doi:10.1155/2009/574398.
- Hagar Zohar, Susan J. Muller. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA // Nanoscale. — 2011-08-01. — Т. 3, вып. 8. — С. 3027—3039. — ISSN 2040-3372. — doi:10.1039/c1nr10280j.
- Marco Wiltgen, Gernot P. Tilz. DNA microarray analysis: principles and clinical impact // Hematology (Amsterdam, Netherlands). — 2007-08-01. — Т. 12, вып. 4. — С. 271—287. — ISSN 1607-8454. — doi:10.1080/10245330701283967.
- pubmeddev. Comparison of fluorescent tag DNA labeling methods used for expression - PubMed - NCBI. www.ncbi.nlm.nih.gov. Дата обращения: 13 апреля 2017.
- Roger Bumgarner. Overview of DNA microarrays: types, applications, and their future // Current Protocols in Molecular Biology. — 2013-01-01. — Т. Chapter 22. — С. Unit 22.1.. — ISSN 1934-3647. — doi:10.1002/0471142727.mb2201s101.
- M. J. Solomon, P. L. Larsen, A. Varshavsky. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene (англ.) // Cell. — Cell Press, 1988-06-17. — Vol. 53, iss. 6. — P. 937—947. — ISSN 0092-8674.
- Christine E. Horak, Michael Snyder. ChIP-chip: a genomic approach for identifying transcription factor binding sites // Methods in Enzymology. — 2002-01-01. — Т. 350. — С. 469—483. — ISSN 0076-6879.
- Michael J. Buck, Jason D. Lieb. ChIP-chip: considerations for the design, analysis, and application of genome-wide chromatin immunoprecipitation experiments // Genomics. — Academic Press, 2004-03-01. — Т. 83, вып. 3. — С. 349—360. — ISSN 0888-7543.
- D. G. Wang, J. B. Fan, C. J. Siao, A. Berno, P. Young. Large-scale identification, mapping, and genotyping of single-nucleotide polymorphisms in the human genome // Science (New York, N.Y.). — 1998-05-15. — Т. 280, вып. 5366. — С. 1077—1082. — ISSN 0036-8075.
- J. B. Fan, A. Oliphant, R. Shen, B. G. Kermani, F. Garcia. Highly parallel SNP genotyping // Cold Spring Harbor Symposia on Quantitative Biology. — 2003-01-01. — Т. 68. — С. 69—78. — ISSN 0091-7451.
- A. Kurg, N. Tõnisson, I. Georgiou, J. Shumaker, J. Tollett. Arrayed primer extension: solid-phase four-color DNA resequencing and mutation detection technology // Genetic Testing. — 2000-01-01. — Т. 4, вып. 1. — С. 1—7. — ISSN 1090-6576. — doi:10.1089/109065700316408.
- Kevin L. Gunderson, Frank J. Steemers, Hongi Ren, Pauline Ng, Lixin Zhou. Whole-genome genotyping // Methods in Enzymology. — 2006-01-01. — Т. 410. — С. 359—376. — ISSN 0076-6879. — doi:10.1016/S0076-6879(06)10017-8.
- Paul J. Gardina, Tyson A. Clark, Brian Shimada, Michelle K. Staples, Qing Yang. Alternative splicing and differential gene expression in colon cancer detected by a whole genome exon array // BMC genomics. — 2006-12-27. — Т. 7. — С. 325. — ISSN 1471-2164. — doi:10.1186/1471-2164-7-325.
- John Castle, Phil Garrett-Engele, Christopher D. Armour, Sven J. Duenwald, Patrick M. Loerch. Optimization of oligonucleotide arrays and RNA amplification protocols for analysis of transcript structure and alternative splicing (англ.) // BioMed Central. — 2003-01-01. — Vol. 4, iss. 10. — P. R66. — ISSN 1474-760X. — doi:10.1186/gb-2003-4-10-r66.
- Weerayuth Kittichotirat, Roger E. Bumgarner, Sirkka Asikainen, Casey Chen. Identification of the pangenome and its components in 14 distinct Aggregatibacter actinomycetemcomitans strains by comparative genomic analysis // PloS One. — 2011-01-01. — Т. 6, вып. 7. — С. e22420. — ISSN 1932-6203. — doi:10.1371/journal.pone.0022420.
