Микробиом
Микробиом (micro — «маленький», bios — «жизнь») — сообщество микроорганизмов, населяющих конкретную среду обитания, или совокупность генов микроорганизмов такого сообщества[1]. Термин часто используется как синоним «микробиоты» или «микрофлоры»[1], впервые был употреблён в статье 1952 года, посвящённой загрязнению воды стоками из канализации[2]. Собственным микробным сообществом (микробиомом) обладают все экосистемы, начиная от тканей и органов отдельных организмов[3] и заканчивая целыми средами обитания[4]. Микробиом участвует в важнейших экосистемных процессах, способствуя как метаболизму хозяина в локальном масштабе[5], так и биогеохимическому круговороту питательных веществ в глобальном[6].
Самыми генетически разнообразными являются почвенные микробиомы, которые влияют на взаимодействие между наземными и подземными биомами, водными экосистемами и земной атмосферой. Микробиом океана оказывает влияние на климат планеты, участвует в круговороте азота и других питательных веществ.
Микробиомы сред обитания
Микробиомы почв
Почвенные микробиомы — самые генетически разнообразные экосистемы на Земле, включающие сложные комплексы бактерий, архей и эукариот[7]. Почва содержит около 108—1010 клеток микроорганизмов на грамм[8], что многократно превышает их содержание на миллилитр (104—107 клеток) в Мировом океане[9]. Почвенные микроорганизмы играют важнейшую роль в поддержании геохимического состава биосферы[9]. Они ответственны за циркуляцию биогенных элементов (азота, углерода и фосфора и других), их преобразование в доступный для растений вид, поддержание структуры почвы[10]. Микроорганизмы фиксируют атмосферный азот и углерод, производят органические вещества и иммобилизуют достаточное количество азота и других питательных веществ, чтобы инициировать процессы круговорота азота в молодой почве[10]. Почва представляет собой резервуар видов микроорганизмов, которые напрямую поддерживают (полезные симбионты) или препятствуют (патогены) росту растений, а также изменяют биодоступность питательных веществ и токсинов[11]. Микробиом почв влияет на взаимодействие между наземными и подземными биомами, водными экосистемами и земной атмосферой, обеспечивает обмен между геологическими запасами и биосферой[12].
Среди наиболее важных представителей почвенного микробиома, вносящих наибольший вклад в формирование почвенного микробного сообщества — бактерии родов Pseudomonas (производят различные антибиотики, литические ферменты, гормоны растений, этилен, ауксины и гиббереллины), Azotobacter, Clostridium, Rhizobium и Bradyrhizobium (усваивают атмосферный азот)[13], а также актиномицеты (Streptomyces) и грибы — Mycorrhizae[14].
Океаны и моря
Океанический микробиом — это сильно разбавленная микробная система, которая покрывает большую часть поверхности Земли. Его границы простираются от полярных регионов Арктики и Антарктики до кипящих гидротермальных источников в морских глубинах и известковых илов[15]. В совокупности на океанический микробиом приходится ~ 0,0001 % объёма морской воды[16]. Микробиом океана оказывает влияние на климат нашей планеты, участвует в круговороте азота и других питательных веществ. Одно из важнейших свойств океанической микробиоты — наличие в ней первичных продуцентов. В отличие от других микробиомов, в океаническом присутствуют организмы, способные к преобразованию световой энергии, что вносит существенный вклад в циркуляцию энергии по земной экосистеме[17]. Примерное содержание микроорганизмов в Мировом океане варьирует от 104 до 107 клеток на миллилитр в зависимости от глубины, а в озёрах в среднем 106 клеток на миллилитр[9].
Наиболее распространённые представители микробиома океана — бактерии Vibrio, Pelagibacter ubique, Prochlorococcus, Cyanobacteria[18]; а также археи Halobacteria, Haloquadratum walsbyi[19], Pyrolobus fumarii.
Реки
Отличительной чертой экосистемы реки является изменчивость из-за течений. В связи с этим, вдоль течения реки наблюдаются изменения в разнообразии микробиома, которые могут быть постепенными в местах со слабыми движениями водных масс, или резкими, происходящими на стыках с другими экосистемами[20]. Разложение органических веществ, продуцирование парниковых газов, эвтрофикация, усвоение металлических загрязнителей, разложение ксенобиотических соединений — это лишь малая часть процессов, осуществляемых речным микробиомом[20]. Особое практическое значение имеет, что у членов микробиома загрязнённых рек выявляются гены, способные расщеплять различные токсины и ксенобиотики. Учитывая, что реки являются источниками питьевой воды, данные изменения могут отражаться на человеке и животных[21].
Наиболее распространённые представители микробиомов рек — бактерии Actinobacteria, Betaproteobacteria, Flavobacteriia[22].
Микробиомы живых организмов
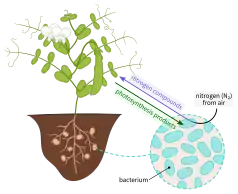
Микробиомы растений
Микроорганизмы различаются по типу взаимоотношений с растением, например, встречаются патогены, эндофиты и симбионты[23]. Потребности растений в азоте, фосфоре и железе удовлетворяются за счёт активности микроорганизмов в почве, на её поверхности и в непосредственной близости от неё[24]. В свою очередь, источником углерода для представителей растительного микробиома могут служить как исключительно корневые экссудаты растений (например, род Myxococcus), так и органическое вещество почвы (например, отряд Sphingomonadales)[25]. Почвенные микроорганизмы колонизируют корни растений. Комменсальные, симбиотические или патогенные бактерии и грибы надземных частей растений, также частично происходят из корней и почвы. На формирование микробного сообщества в ризосфере оказывают влияние генотип растения-хозяина, тип почвы и методы выращивания[26], изменяют фенологию и время цветения[27], влияют на выработку сухого вещества побегов растений[28] и вызывают системную устойчивость к болезням[29]. Также вероятно, что микробиомы влияют на качество смол, фруктов, мёда и эфирных масел[30].
Микробиомы животных
Микробиомами обладают практически все живые организмы: начиная от губок[31] и заканчивая человеком. Наличие и состав резидентных микробных сообществ в организме животного напрямую влияет на его физиологическое состояние[32]. Микробные сообщества наиболее часто заселяют пищеварительную систему и внешнюю поверхность тела животных[33][34], а также репродуктивные органы[35] и ротовую полость[36] человека. Помимо этого, микроорганизмы можно обнаружить и в кровеносной системе человека[37] и даже в гемолимфе насекомых[38]. У некоторых животных есть специализированные органы, в которых обитают определённые группы микроорганизмов[39]. При этом ассоциации между животными и конкретными видами не случайны[40]. В организме хозяина формируются комплексные приспособления для обеспечения полезных микроорганизмов пищей, подходящей средой обитания и защитой от других представителей[41].
Хорошо известно, что микробиом может участвовать в процессах пищеварения, расщепляя соединения, недоступные для собственных ферментативных систем хозяина, что расширяет животным кормовую базу[42]. Микроорганизмы способны нейтрализовать токсины, синтезировать различные молекулы, необходимые для метаболизма хозяина (витамины и др.)[43]. Микробы, населяющие поверхность тела, защищают хозяина от патогенов. Возможно, микроорганизмы оказывают влияние не только на физиологическое состояние животного, но и на его поведение, путём синтеза сигнальных молекул[38].
Методы изучения микробиома
Таргетное секвенирование ампликонов
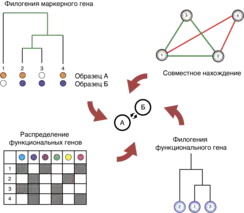
Для определения таксономического разнообразия в изучаемом образце используется секвенирование гена 16S рибосомальной РНК (16S рРНК), присутствующей во всех живых организмах. Вместе с тем, использование 16S рРНК в качестве индикатора разнообразия зачастую не позволяет различить виды из-за отсутствия у них различий в этом гене[44]. Микробное сообщество часто определяется стабильным видовым составом, но такая стабильность не всегда соблюдается при многократном заборе проб (микробное сообщество не гомогенно на всём своём протяжении, его состав также может колебаться с течением времени)[45].
После секвенирования ампликонов для определения состава микробного сообщества используются методы молекулярной филогении. Это делается путём кластеризации ампликонов в оперативные таксономические единицы (OTU) и выведения филогенетических связей между последовательностями ДНК[46].
Метагеномное секвенирование
Альтернативой исследованиям маркерных генов (16S рРНК) является метагеномный анализ с помощью секвенирования методом дробовика. Получаемый набор последовательностей может охватить весь геном отобранных микроорганизмов, а не только отдельный маркерный ген. Такой подход может выявить функциональную комплиментарность внутри изучаемого образца и на основе этого предложить взаимодействия между его членами. Однако сборка и присвоение функции и таксономии метагеномным последовательностям — сложная задача, которая часто порождает множество предсказаний с низкой достоверностью[47]. Часто также возникает неоднозначность относительно того, каким именно организмам принадлежит конкретно взятая последовательность, так как не всегда доступен полный геном организма[48].
Метапротеомика и метатранскриптомика
Помимо изучения геномов, особый интерес представляют транскриптомы и протеомы микроорганизмов в составе сообществ. Комплексное изучение молекул белка и молекул РНК в составе образца, содержащего в себе представителей различных видов бактерий, называются соответственно метапротеомикой и метатранскриптомикой. Эти подходы, в отличие от описанных выше, позволяют не просто оценить генетический потенциал сообщества, а получить представление об активных генах и синтезируемых молекулах белка и метаболитах[49].
Для метатранскриптомных исследований обычно проводится пиросеквенирование РНК, выделенной из всего сообщества[50]. Данные для метапротеомики получают выделением белков из клеток сообщества с их последующим анализом методом масс-спектрометрии[49].
Определение взаимодействий внутри микробиома
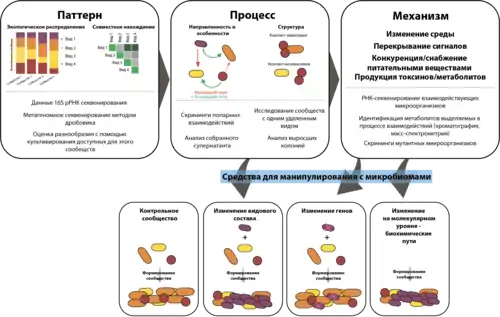
Существует трёхфазный подход для выявления причин и последствий микробных взаимодействий[51]. Он основан на поиске паттернов в видовом составе микробных сообществ, изучении роли отдельных видов в функционировании всего сообщества, поиске конкретных механизмов, обуславливающих эту роль.
Для выявления совместной встречаемости видов в составе сообществ, которые могут быть объяснены взаимодействиями внутри них, используются метагеномные исследования in situ. Для изучения межвидовых взаимодействий в составе сообществ, используются экспериментальные микробные сообщества: изучается изменение фенотипа сообщества в попарных скринингах или при удалении отдельного вида из сообщества для установлении его роли в нём. Для идентификации генетических и молекулярных механизмов, лежащих в основе этих микробных взаимодействий, используются транскриптомные, метаболомные и скрининги, а также исследуются генетически модифицированные микроорганизмы[52].
Влияние взаимодействий внутри микробиома на эволюцию микроорганизмов
Эволюционные исследования показали, что некоторые характеристики микроорганизмов, включая метаболизм, стрессоустойчивость и вирулентность, могут быстро эволюционировать в популяции по мере последовательной смены поколений[53]. Взаимодействия между микроорганизмами могут ингибировать фенотипическую и генетическую эволюцию конкретного вида путём сокращения размеров популяции и уменьшения потенциальной генетической изменчивости для отбора, или могут способствовать адаптации, когда результатом взаимодействия является смена экологической ниши эволюционирующего вида. Исследования нескольких представителей рода Pseudomonas показали, что конкуренция со стороны других видов может как ингибировать[54], так и стимулировать их эволюцию[55]. Ещё одним примером может служить появление в микробиоме функциональной специализации у микроорганизмов, часто являющейся результатом эндосимбиоза. Так, ассоциация между бактериями Candidatus «Moranella endobia» и Candidatus «Tremblaya princeps», живущими внутри клеток червеца цитрусового (Planococcus citri), привела к разделению между ними промежуточных звеньев путей синтеза фенилаланина, аргинина и изолейцина[56].
Экологическое перекрывание или эквивалентность могут лежать в основе часто наблюдаемых таксономических различий между образцами, взятыми из одних и тех же или похожих мест обитания. Наиболее убедительным примером этого является отсутствие постоянного «основного» микробиома во многих органах человека[57]. Так, S.M. Huse et al. (2012) в исследовании, проведённом в рамках международного проекта «Микробиом человека»[58], показали, что чёткая категоризация микробиоты фактически невозможна и правильнее говорить не о существовании энтеротипов, а о наличии непрерывного градиента микробных сообществ[59][60]. Проект «Микробиом человека» появился в 2007 году, и исследования в этой сфере проводились Национальными институтами здравоохранения США вплоть до 2016 года. Его целью было получить наиболее полную картину разнообразия микрофлоры различных органов человека[61].
Хотя не представляется возможным выявить «основной кишечный» микробиом человека, все же при более точном определении можно охарактеризовать основной микробом «здорового толстого кишечника с высоким содержанием белка и жиров животного происхождения». Преемственность может также играть роль, например, при детерминировании зубного налёта: один и тот же участок может быть занят «ранними» или «поздними» сообществами, которые возникают после нарушения[5]. Также при исследовании распределения операционных таксономических единиц в микробиомах почвы, озёрной воды и солёных отложений было выявлено отсутствие перекрывания между ними даже при уменьшении порога идентичности последовательностей до 89 %[62].
Модифицирование микробиома
В последние годы изучение микробиома быстро прогрессировало от поиска взаимосвязей между хозяином и микробиомом до понимания его роли в развитии заболеваний. Манипулирование естественной микробной флорой человека открыло уникальную возможность для повышения эффективности комменсальной флоры и снижения рисков возникновения с ней связанных заболеваний. Появились микробно-центрированные терапевтические стратегии, которые обеспечивают или воссоздают желаемую функцию интересующего микробного сообщества, либо подавляют или устраняют определённый нежелательный или патогенный элемент[63]. Например, бактериотерапия была успешно использована при инфекциях Clostridium difficile за счёт пересадки фекальных микробиомов от здоровых доноров пациенту, что привело к восстановлению нормальной микробиоты[64]. В других случаях, благодаря методам системной биологии идентифицировали отдельных микроорганизмов и некоторые их консорциумы, которые могут, в значительной степени, повторять эффекты пересадки фекальных микробиомов[65].
Также существуют экспериментальные методики модификации микробиома in situ. Для этих целей используются CRISPR/Cas9 системы. Они способные узнавать определённые участки генома патогенов, отвечающие за устойчивость к антибиотикам, и приводить к их деградации. Для доставки этих систем в бактериальные клетки используются бактериофаги. При этом CRISPR/Cas9 нуклеазы, благодаря специфичности своего устройства, поражают только определённые штаммы потенциально опасных бактерий. В связи с этим, их использование безопасно для нормальной микрофлоры и может предотвращать развитие дисбактериоза[66].
Проекты
Помимо выше затронутого проекта «Микробиом человека», существует также созданный в 2010 году проект «Микробиом Земли» — это инициатива по сбору природных образцов и анализу микробного сообщества по всему миру. Микроорганизмы очень распространены, разнообразны и играют важную роль в экологической системе[6]. Тем не менее, по состоянию на 2010 г. было подсчитано, что в окружающей среде отсеквенировано менее 1 % от общей ДНК, обнаруженной в литрах морской воды или грамме почвы[67]. А конкретные взаимодействия между микроорганизмами остаются неизвестными. Цель проекта «Микробиом Земли» состоит в том, чтобы обработать до 200 000 образцов в различных биомах, создав полную базу данных микроорганизмов на Земле, чтобы охарактеризовать окружающую среду и экосистемы по микробному составу и взаимодействию. Используя эти данные, можно предложить и протестировать новые экологические и эволюционные теории[68].
Примечания
- Microbiome : [англ.] // Merriam-Webster.com Dictionary. — Merriam-Webster. — Дата обращения: 12.04.2020.
- Mohr JL. Protozoa as indicators of pollution. (англ.) // The Scientific Monthly. — 1952.
- E. K. Costello, K. Stagaman, L. Dethlefsen, B. J. M. Bohannan, D. A. Relman. The Application of Ecological Theory Toward an Understanding of the Human Microbiome // Science. — 2012-06-06. — Т. 336, вып. 6086. — С. 1255–1262. — ISSN 1095-9203 0036-8075, 1095-9203. — doi:10.1126/science.1224203.
- Sean M Gibbons, Jack A Gilbert. Microbial diversity — exploration of natural ecosystems and microbiomes // Current Opinion in Genetics & Development. — 2015-12. — Т. 35. — С. 66–72. — ISSN 0959-437X. — doi:10.1016/j.gde.2015.10.003.
- Dirk Gevers, Rob Knight, Joseph F. Petrosino, Katherine Huang, Amy L. McGuire. The Human Microbiome Project: A Community Resource for the Healthy Human Microbiome // PLoS Biology. — 2012-08-14. — Т. 10, вып. 8. — С. e1001377. — ISSN 1545-7885. — doi:10.1371/journal.pbio.1001377.
- P. G. Falkowski, T. Fenchel, E. F. Delong. The Microbial Engines That Drive Earth's Biogeochemical Cycles // Science. — 2008-05-23. — Т. 320, вып. 5879. — С. 1034–1039. — ISSN 1095-9203 0036-8075, 1095-9203. — doi:10.1126/science.1153213.
- T. P. Curtis, W. T. Sloan, J. W. Scannell. Estimating prokaryotic diversity and its limits (англ.) // Proceedings of the National Academy of Sciences. — 2002-08-06. — Vol. 99, iss. 16. — P. 10494–10499. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.142680199.
- Xavier Raynaud, Naoise Nunan. Spatial Ecology of Bacteria at the Microscale in Soil (англ.) // PLoS ONE / Francesco Pappalardo. — 2014-01-28. — Vol. 9, iss. 1. — P. e87217. — ISSN 1932-6203. — doi:10.1371/journal.pone.0087217.
- W. B. Whitman, D. C. Coleman, W. J. Wiebe. Prokaryotes: The unseen majority // Proceedings of the National Academy of Sciences. — 1998-06-09. — Т. 95, вып. 12. — С. 6578–6583. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.95.12.6578.
- Anamika Dubey, Muneer Ahmad Malla, Farhat Khan, Kanika Chowdhary, Shweta Yadav. Soil microbiome: a key player for conservation of soil health under changing climate // Biodiversity and Conservation. — 2019-04-04. — Т. 28, вып. 8—9. — С. 2405–2429. — ISSN 1572-9710 0960-3115, 1572-9710. — doi:10.1007/s10531-019-01760-5.
- Mm Roper, V Gupta. Management-practices and soil biota (англ.) // Soil Research. — 1995. — Vol. 33, iss. 2. — P. 321. — ISSN 1838-675X. — doi:10.1071/SR9950321.
- Kate H. Orwin, Bryan A. Stevenson, Simeon J. Smaill, Miko U. F. Kirschbaum, Ian A. Dickie. Effects of climate change on the delivery of soil-mediated ecosystem services within the primary sector in temperate ecosystems: a review and New Zealand case study (англ.) // Global Change Biology. — 2015-08. — Vol. 21, iss. 8. — P. 2844–2860. — doi:10.1111/gcb.12949.
- Satyavir Singh Sind, Sita Ram Choudh. Suppression of Rhizoctonia solani Root Rot Disease of Clusterbean (Cyamopsis tetragonoloba) and Plant Growth Promotion by Rhizosphere Bacteria // Plant Pathology Journal. — 2015-02-01. — Т. 14, вып. 2. — С. 48–57. — ISSN 1812-5387. — doi:10.3923/ppj.2015.48.57.
- N. S. Subba Rao. Soil Microbiology (Fourth Edition of Soil Microorganisms and Plant Growth). — Oxford and IBH Publishing Company Pvt. Limited, 2005. — 407 с.
- Michael Pester, Christa Schleper, Michael Wagner. The Thaumarchaeota: an emerging view of their phylogeny and ecophysiology // Current Opinion in Microbiology. — 2011-06. — Т. 14, вып. 3. — С. 300–306. — ISSN 1369-5274. — doi:10.1016/j.mib.2011.04.007.
- J. A. Fuhrman, J. A. Steele, I. Hewson, M. S. Schwalbach, M. V. Brown. A latitudinal diversity gradient in planktonic marine bacteria // Proceedings of the National Academy of Sciences. — 2008-05-28. — Т. 105, вып. 22. — С. 7774–7778. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.0803070105.
- Yinon M. Bar-On, Rob Phillips, Ron Milo. The biomass distribution on Earth // Proceedings of the National Academy of Sciences. — 2018-05-21. — Т. 115, вып. 25. — С. 6506–6511. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.1711842115.
- Ricardo Beiras. Chapter 16 - Biological Tools for Monitoring: Biomarkers and Bioassays (англ.) // Marine Pollution / Ricardo Beiras. — Elsevier, 2018-01-01. — P. 265–291. — ISBN 978-0-12-813736-9. — doi:10.1016/b978-0-12-813736-9.00016-7.
- W Stoeckenius. Walsby's square bacterium: fine structure of an orthogonal procaryote. (англ.) // Journal of Bacteriology. — 1981. — Vol. 148, iss. 1. — P. 352–360. — ISSN 1098-5530 0021-9193, 1098-5530. — doi:10.1128/JB.148.1.352-360.1981.
- Domenico Savio, Lucas Sinclair, Umer Z. Ijaz, Philipp Stadler, Alfred P. Blaschke, Georg H. Reischer. Bacterial diversity along a 2 600 km river continuum. dx.doi.org (7 октября 2014). Дата обращения: 24 марта 2020.
- F.F Reinthaler, J Posch, G Feierl, G Wüst, D Haas. Antibiotic resistance of E. coli in sewage and sludge // Water Research. — 2003-04. — Т. 37, вып. 8. — С. 1685–1690. — ISSN 0043-1354. — doi:10.1016/s0043-1354(02)00569-9.
- Caroline S Fortunato, Alexander Eiler, Lydie Herfort, Joseph A Needoba, Tawnya D Peterson. Determining indicator taxa across spatial and seasonal gradients in the Columbia River coastal margin (англ.) // The ISME Journal. — 2013-10. — Vol. 7, iss. 10. — P. 1899–1911. — ISSN 1751-7370 1751-7362, 1751-7370. — doi:10.1038/ismej.2013.79.
- Mónica Rosenblueth, Esperanza Martínez-Romero. Bacterial Endophytes and Their Interactions with Hosts // Molecular Plant-Microbe Interactions. — 2006-08. — Т. 19, вып. 8. — С. 827–837. — ISSN 0894-0282. — doi:10.1094/mpmi-19-0827.
- Ben Lugtenberg, Faina Kamilova. Plant-Growth-Promoting Rhizobacteria // Annual Review of Microbiology. — 2009-10. — Т. 63, вып. 1. — С. 541–556. — ISSN 1545-3251 0066-4227, 1545-3251. — doi:10.1146/annurev.micro.62.081307.162918.
- Feth el Zahar Haichar, Christine Marol, Odile Berge, J Ignacio Rangel-Castro, James I Prosser. Plant host habitat and root exudates shape soil bacterial community structure // The ISME Journal. — 2008-08-28. — Т. 2, вып. 12. — С. 1221–1230. — ISSN 1751-7370 1751-7362, 1751-7370. — doi:10.1038/ismej.2008.80.
- Gaston Zolla, Dayakar V. Badri, Matthew G. Bakker, Daniel K. Manter, Jorge M. Vivanco. Soil microbiomes vary in their ability to confer drought tolerance to Arabidopsis (англ.) // Applied Soil Ecology. — 2013-06. — Vol. 68. — P. 1–9. — doi:10.1016/j.apsoil.2013.03.007.
- Kevin Panke-Buisse, Angela C Poole, Julia K Goodrich, Ruth E Ley, Jenny Kao-Kniffin. Selection on soil microbiomes reveals reproducible impacts on plant function (англ.) // The ISME Journal. — 2015-04. — Vol. 9, iss. 4. — P. 980–989. — ISSN 1751-7370 1751-7362, 1751-7370. — doi:10.1038/ismej.2014.196.
- Susanne Schreiter, Guo-Chun Ding, Holger Heuer, Günter Neumann, Martin Sandmann. Effect of the soil type on the microbiome in the rhizosphere of field-grown lettuce // Frontiers in Microbiology. — 2014-04-08. — Т. 5. — ISSN 1664-302X. — doi:10.3389/fmicb.2014.00144.
- Santosh Babu, Ngangom Bidyarani, Preeti Chopra, Dilip Monga, Rishi Kumar. Evaluating microbe-plant interactions and varietal differences for enhancing biocontrol efficacy in root rot disease challenged cotton crop (англ.) // European Journal of Plant Pathology. — 2015-06. — Vol. 142, iss. 2. — P. 345–362. — ISSN 1573-8469 0929-1873, 1573-8469. — doi:10.1007/s10658-015-0619-6.
- Günter Brader, Stéphane Compant, Birgit Mitter, Friederike Trognitz, Angela Sessitsch. Metabolic potential of endophytic bacteria (англ.) // Current Opinion in Biotechnology. — 2014-06. — Vol. 27. — P. 30–37. — doi:10.1016/j.copbio.2013.09.012.
- J. Pamela Engelberts, Steven J. Robbins, Jasper M. de Goeij, Manuel Aranda, Sara C. Bell. Characterization of a sponge microbiome using an integrative genome-centric approach (англ.) // The ISME Journal. — 2020-01-28. — ISSN 1751-7370 1751-7362, 1751-7370. — doi:10.1038/s41396-020-0591-9.
- Delaney L Miller, Audrey J Parish, Irene LG Newton. Transitions and transmission: behavior and physiology as drivers of honey bee-associated microbial communities (англ.) // Current Opinion in Microbiology. — 2019-08-01. — Vol. 50. — P. 1–7. — ISSN 1369-5274. — doi:10.1016/j.mib.2019.08.001.
- D. C. Woodhams, L. A. Rollins-Smith, R. A. Alford, M. A. Simon, R. N. Harris. Innate immune defenses of amphibian skin: antimicrobial peptides and more // Animal Conservation. — 2007-11. — Т. 10, вып. 4. — С. 425–428. — ISSN 1469-1795 1367-9430, 1469-1795. — doi:10.1111/j.1469-1795.2007.00150.x.
- Elizabeth A. Grice, Julia A. Segre. The skin microbiome (англ.) // Nature Reviews Microbiology. — 2011-04. — Vol. 9, iss. 4. — P. 244–253. — ISSN 1740-1534 1740-1526, 1740-1534. — doi:10.1038/nrmicro2537.
- Inmaculada Moreno, Carlos Simon. Deciphering the effect of reproductive tract microbiota on human reproduction // Reproductive Medicine and Biology. — 2019-01. — Т. 18, вып. 1. — С. 40–50. — ISSN 1445-5781. — doi:10.1002/rmb2.12249.
- M. Kilian, I. L. C. Chapple, M. Hannig, P. D. Marsh, V. Meuric. The oral microbiome – an update for oral healthcare professionals (англ.) // British Dental Journal. — 2016-11. — Vol. 221, iss. 10. — P. 657–666. — ISSN 1476-5373. — doi:10.1038/sj.bdj.2016.865.
- Stefan Panaiotov, Georgi Filevski, Michele Equestre, Elena Nikolova, Reni Kalfin. Cultural Isolation and Characteristics of the Blood Microbiome of Healthy Individuals // Advances in Microbiology. — 2018. — Т. 08, вып. 05. — С. 406–421. — ISSN 2165-3410 2165-3402, 2165-3410. — doi:10.4236/aim.2018.85027.
- Jialei Xie, Igor Vilchez, Mariana Mateos. Spiroplasma Bacteria Enhance Survival of Drosophila hydei Attacked by the Parasitic Wasp Leptopilina heterotoma // PLoS ONE. — 2010-08-13. — Т. 5, вып. 8. — С. e12149. — ISSN 1932-6203. — doi:10.1371/journal.pone.0012149.
- Holly L. Lutz, S. Tabita Ramírez-Puebla, Lisa Abbo, Amber Durand, Cathleen Schlundt, Neil Gottel. A simple microbiome in the European common cuttlefish, Sepia officinalis. dx.doi.org (11 октября 2018). Дата обращения: 6 апреля 2020.
- Stéphane Hacquard, Ruben Garrido-Oter, Antonio González, Stijn Spaepen, Gail Ackermann. Microbiota and Host Nutrition across Plant and Animal Kingdoms (англ.) // Cell Host & Microbe. — 2015-05. — Vol. 17, iss. 5. — P. 603–616. — doi:10.1016/j.chom.2015.04.009.
- Heather L. Eisthen, Kevin R. Theis. Animal–microbe interactions and the evolution of nervous systems // Philosophical Transactions of the Royal Society B: Biological Sciences. — 2016-01-05. — Т. 371, вып. 1685. — ISSN 0962-8436. — doi:10.1098/rstb.2015.0052.
- Margaret McFall-Ngai, Michael G. Hadfield, Thomas C. G. Bosch, Hannah V. Carey, Tomislav Domazet-Lošo. Animals in a bacterial world, a new imperative for the life sciences (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2013-02-26. — Vol. 110, iss. 9. — P. 3229. — doi:10.1073/pnas.1218525110.
- Paul Baumann. Biology bacteriocyte-associated endosymbionts of plant sap-sucking insects // Annual Review of Microbiology. — 2005. — Т. 59. — С. 155–189. — ISSN 0066-4227. — doi:10.1146/annurev.micro.59.030804.121041.
- Duccio Medini, Claudio Donati, Hervé Tettelin, Vega Masignani, Rino Rappuoli. The microbial pan-genome (англ.) // Current Opinion in Genetics & Development. — 2005-12. — Vol. 15, iss. 6. — P. 589–594. — doi:10.1016/j.gde.2005.09.006.
- J. G. Caporaso, C. L. Lauber, W. A. Walters, D. Berg-Lyons, C. A. Lozupone. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample // Proceedings of the National Academy of Sciences. — 2010-06-03. — Т. 108, вып. Supplement_1. — С. 4516–4522. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.1000080107.
- J Gregory Caporaso, Justin Kuczynski, Jesse Stombaugh, Kyle Bittinger, Frederic D Bushman. QIIME allows analysis of high-throughput community sequencing data (англ.) // Nature Methods. — 2010-05. — Vol. 7, iss. 5. — P. 335–336. — ISSN 1548-7105 1548-7091, 1548-7105. — doi:10.1038/nmeth.f.303.
- T. Prakash, T. D. Taylor. Functional assignment of metagenomic data: challenges and applications // Briefings in Bioinformatics. — 2012-07-06. — Т. 13, вып. 6. — С. 711–727. — ISSN 1477-4054 1467-5463, 1477-4054. — doi:10.1093/bib/bbs033.
- Justin Kuczynski, Christian L. Lauber, William A. Walters, Laura Wegener Parfrey, José C. Clemente. Experimental and analytical tools for studying the human microbiome (англ.) // Nature Reviews Genetics. — 2012-01. — Vol. 13, iss. 1. — P. 47–58. — ISSN 1471-0064 1471-0056, 1471-0064. — doi:10.1038/nrg3129.
- Pierre-Alain Maron, Lionel Ranjard, Christophe Mougel, Philippe Lemanceau. Metaproteomics: A New Approach for Studying Functional Microbial Ecology (англ.) // Microbial Ecology. — 2007-05-04. — Vol. 53, iss. 3. — P. 486–493. — ISSN 1432-184X 0095-3628, 1432-184X. — doi:10.1007/s00248-006-9196-8.
- Yanmei Shi, Gene W. Tyson, Edward F. DeLong. Metatranscriptomics reveals unique microbial small RNAs in the ocean’s water column (англ.) // Nature. — 2009-05. — Vol. 459, iss. 7244. — P. 266–269. — ISSN 1476-4687 0028-0836, 1476-4687. — doi:10.1038/nature08055.
- Kastman et al. Biotic Interactions Shape the Ecological Distributions of Staphylococcus Species // mBio. — 2016.
- Casey M Cosetta, Benjamin E Wolfe. Causes and consequences of biotic interactions within microbiomes (англ.) // Current Opinion in Microbiology. — 2019-08-01. — Vol. 50. — P. 35–41. — ISSN 1369-5274. — doi:10.1016/j.mib.2019.09.004.
- Richard E Lenski. Experimental evolution and the dynamics of adaptation and genome evolution in microbial populations // The ISME Journal. — 2017-05-16. — Т. 11, вып. 10. — С. 2181–2194. — ISSN 1751-7370 1751-7362, 1751-7370. — doi:10.1038/ismej.2017.69.
- James P. J. Hall, Ellie Harrison, Michael A. Brockhurst. Competitive species interactions constrain abiotic adaptation in a bacterial soil community // Evolution Letters. — 2018-09-25. — Т. 2, вып. 6. — С. 580–589. — ISSN 2056-3744. — doi:10.1002/evl3.83.
- Quan-Guo Zhang, Richard J. Ellis, H. Charles J. Godfray. THE EFFECT OF A COMPETITOR ON A MODEL ADAPTIVE RADIATION // Evolution. — 2012-01-23. — Т. 66, вып. 6. — С. 1985–1990. — ISSN 0014-3820. — doi:10.1111/j.1558-5646.2011.01559.x.
- John P. McCutcheon, Carol D. von Dohlen. An Interdependent Metabolic Patchwork in the Nested Symbiosis of Mealybugs // Current Biology. — 2011-08. — Т. 21, вып. 16. — С. 1366–1372. — ISSN 0960-9822. — doi:10.1016/j.cub.2011.06.051.
- Susan M. Huse, Yuzhen Ye, Yanjiao Zhou, Anthony A. Fodor. A Core Human Microbiome as Viewed through 16S rRNA Sequence Clusters // PLoS ONE. — 2012-06-13. — Т. 7, вып. 6. — С. e34242. — ISSN 1932-6203. — doi:10.1371/journal.pone.0034242.
- Human Microbiome Project, HMP.
- Susan M. Huse, Yuzhen Ye, Yanjiao Zhou, Anthony A. Fodor. A Core Human Microbiome as Viewed through 16S rRNA Sequence Clusters (англ.) // PLOS ONE. — 2012. — Vol. 7, iss. 6. — P. e34242. — ISSN 1932-6203. — doi:10.1371/journal.pone.0034242.
- Ситкин, С. И., Ткаченко, Е. И., Вахитов, Т. Я. Филометаболическое ядро микробиоты кишечника // Альманах клинической медицины. — 2015). — № 40. — С. 12—34. — doi:10.18786/2072-0505-2015-40-12-34.
- Peter J. Turnbaugh, Ruth E. Ley, Micah Hamady, Claire M. Fraser-Liggett, Rob Knight. The Human Microbiome Project (англ.) // Nature. — 2007-10. — Vol. 449, iss. 7164. — P. 804–810. — ISSN 1476-4687 0028-0836, 1476-4687. — doi:10.1038/nature06244.
- Diana R. Nemergut, Elizabeth K. Costello, Micah Hamady, Catherine Lozupone, Lin Jiang. Global patterns in the biogeography of bacterial taxa // Environmental Microbiology. — 2010-08-01. — Т. 13, вып. 1. — С. 135–144. — ISSN 1462-2912. — doi:10.1111/j.1462-2920.2010.02315.x.
- Travis Whitfill, Julia Oh. Recoding the metagenome: microbiome engineering in situ (англ.) // Current Opinion in Microbiology. — 2019-08-01. — Vol. 50. — P. 28–34. — ISSN 1369-5274. — doi:10.1016/j.mib.2019.09.005.
- Wenjia Hui, Ting Li, Weidong Liu, Chunyan Zhou, Feng Gao. Fecal microbiota transplantation for treatment of recurrent C. difficile infection: An updated randomized controlled trial meta-analysis // PLOS ONE. — 2019-01-23. — Т. 14, вып. 1. — С. e0210016. — ISSN 1932-6203. — doi:10.1371/journal.pone.0210016.
- Luciano Adorini. Faculty of 1000 evaluation for Precision microbiome reconstitution restores bile acid mediated resistance to Clostridium difficile.. F1000 - Post-publication peer review of the biomedical literature (29 ноября 2014). Дата обращения: 24 марта 2020.
- David Bikard, Chad W Euler, Wenyan Jiang, Philip M Nussenzweig, Gregory W Goldberg. Exploiting CRISPR-Cas nucleases to produce sequence-specific antimicrobials (англ.) // Nature Biotechnology. — 2014-11. — Vol. 32, iss. 11. — P. 1146–1150. — ISSN 1546-1696 1087-0156, 1546-1696. — doi:10.1038/nbt.3043.
- Jack A. Gilbert, Folker Meyer, Dion Antonopoulos, Pavan Balaji, C. Titus Brown. Meeting Report: The Terabase Metagenomics Workshop and the Vision of an Earth Microbiome Project (англ.) // Standards in Genomic Sciences. — 2010. — Vol. 3, iss. 3. — P. 243–248. — ISSN 1944-3277. — doi:10.4056/sigs.1433550.
- Jack A Gilbert, Ronald O'Dor, Nicholas King, Timothy M Vogel. The importance of metagenomic surveys to microbial ecology: or why Darwin would have been a metagenomic scientist (англ.) // Microbial Informatics and Experimentation. — 2011-12. — Vol. 1, iss. 1. — P. 5. — ISSN 2042-5783. — doi:10.1186/2042-5783-1-5.