ARNTL
ARNTL (сокр. от англ. Aryl hydrocarbon receptor nuclear translocator-like protein 1) — белок, подобный ядерному транслокатору AHR, кодируется одноимённым геном ARNTL, расположенным на коротком плече (p-плече) 11-ой хромосомы[1]. Также известен как BMAL1, MOP3 и, реже, BHLHE5, BMAL, BMAL1C, JAP3, PASD3 и TIC.
Белок состоит из последовательности 626 аминокислотных остатков и имеет молекулярную массу 68 762 Да[2].
История
Ген ARNTL был первоначально обнаружён в 1997 году двумя группами исследователей: Джоном Б. Хогеншом и сотруд. в марте[3] и Икедой и Номурой в апреле[4], как часть надсемейства факторов транскрипции домена PAS[3]. Было обнаружено, что белок ARNTL, также известный как MOP3, димеризуется с MOP4, CLOCK и факторами, индуцируемые гипоксией[5]. Названия белкам BMAL1 и ARNTL были даны в последующих статьях. Одна из ранних обнаруженных функций белка ARNTL в циркадной регуляции была связана с гетеродимером CLOCK:BMAL1 (CLOCK:ARNTL), который связывался бы через энхансер E-box для активации транскрипции гена, кодирующего гормон вазопрессин[6]. Однако значение гена в циркадных ритмах не было полностью реализовано до тех пор, не был проведён пока нокаут гена у мышей и не продемонстрировал полную потерю циркадных ритмов в локомоции и других видах поведения[7].
Структура
Белок BMAL1 содержит четыре домена в соответствии с его кристаллографической структурой: домен bHLH, два домена PAS, называемые PAS-A и PAS-B, и транс-активирующий домен. Димеризация белков CLOCK: BMAL1 включает сильные взаимодействия между доменами bHLH, PAS-A и PAS-B как CLOCK, так и BMAL1 и образует асимметричный гетеродимер с тремя различными белковыми интерфейсами. Взаимодействие PAS-A между CLOCK и BMAL1, состоит из α-спирали CLOCK PAS-A и ß-листа BMAL1 PAS-A и α-спирального мотива домена BMAL1 PAS-A и ß-листа CLOCK PAS-A[8]. Области CLOCK и BMAL1 PAS-B располагаются параллельно, что приводит к сокрытию различных гидрофобных остатков аминокислот на ß-листе BMAL1 PAS-B и спиральной поверхности CLOCK PAS-B, таких как остатки Tyr 310 и Phe 423[8]. Ключевые взаимодействия со специфическими аминокислотными остатками, особенно CLOCK His 84 и BMAL1 Leu 125, важны для димеризации этих молекул[9].
Функции
Белок, кодируемый геном Bmal1 у млекопитающих, связывается со вторым белком bHLH-PAS через домен PAS, CLOCK (или его паралог, NPAS2) с образованием гетеродимера в клеточном ядре[10]. Через свой домен BHLH этот гетеродимер связывается с элементами отклика E-box[11] в областях промотора Per (Per1 и Per2) и Cry генов (Cry1 и Cry2)[11]. Это связывание активирует транскрипцию и трансляцию белков PER1, PER2, CRY1 и CRY2.
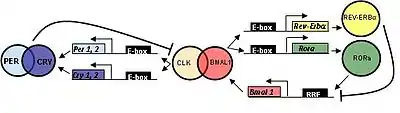
После того, как белки PER и CRY накапливаются до достаточных концентраций, они взаимодействуют со своими мотивам PAS с образованием большого репрессорного комплекса, который перемещается в ядро, чтобы ингибировать транскрипционную активность гетеродимера CLOCK:BMAL1[12]. Этот процесс приводит к ингибированию транскрипции Per и Cry гены, и вызывает снижение концентрации белка PER и CRY. Эта транскрипция, петля отрицательной обратной связи трансляции (TTFL) модулируется в цитоплазме путём фосфорилирования белков PER казеинкиназой 1ε или δ (CK1 ε или CK1 δ), сигнализируя эти белки для деградации протеасомами 26S[11][13]. SIRT1 также регулирует деградацию белка PER посредством ингибирования транскрипционной активности гетеродимера BMAL1:CLOCK путем циркадного (кругового) деацетилирования[14]. Деградация PER-белков предотвращает образование комплекса больших белков и, таким образом, предотвращает ингибирование транскрипционной активности гетеродимера BMAL1:CLOCK[11]. Белок CRY также сигнализируют о деградации поли-убиквированием белка FBXL3, также предотвращая ингибирование гетеродимера CLOCK:BMAL1. Это позволяет возобновить транскрипцию генов Per и Cry. В петле TTFL ночных мышей наблюдалось, что уровни транскрипции пика гена Bmal1 в CT18 во время средней субъективной ночи имеют антифазный уровень транскрипции Per, Cry и других генов контроля частоты, пик в CT6, в середине-субъективного дня. Этот процесс происходит с длиной периода около 24 часов и поддерживает представление о том, что этот молекулярный механизм ритмичен[15].
Регулирование активности Bmal1
В дополнение к циркадной регуляторной TTFL-петле, описанной выше, транскрипция Bmal1 регулируется конкурентным связыванием с сайтом связывания с ретиноидной кислотой, связанным с сайтом-рецептором (RORE) в промоторной области Bmal1. Гетеродимер CLOCK:BMAL1 также связывается с элементами E-box в промоторных областях генов Rev-Erbα и RORα/ß, активируя транскрипцию и трансляцию белков REV-ERB и ROR. Белки REV-ERBα и ROR регулируют экспрессию BMAL1 через вторичный контур обратной связи и конкурируют за связывание с элементами ответа Rev-Erb/ROR в промоторной области Bmal1, в результате чего экспрессию BMAL1 подавляют REV-ERBα и активируют белки ROR. Было показано, что другие ядерные рецепторы тех же семейств (NR1D2 (Rev-erb-β), NR1F2 (ROR-β) и NR1F3 (ROR-γ)) также действуют на транскрипционную активность Bmal1[16][17][18][19].
Несколько посттрансляционных модификаций BMAL1 определяют время циклов обратной связи CLOCK:BMAL1. Фосфорилирование BMAL1 нацеливает его на убиквитинирование и деградацию, а также на деубиквитинирование и стабилизацию. Ацетилирование BMAL1 рекрутирует CRY1 для подавления трансактивации CLOCK:BMAL1[20]. Сумоилирование BMAL1 с помощью небольшого модификатора, связанного с убиквитином 3 (SUMO3), сигнализирует о его убиквитинировании в ядре, что приводит к трансактивации гетеродимера CLOCK:BMAL1[21]. Трансактивация комплекса CLOCK:BMAL1[22] активируется фосфорилированием казеинкиназы 1ε и ингибируется фосфорилированием MAPK[23]. Фосфорилирование CK2α регулирует внутриклеточную локализацию BMAL1[24], а фосфорилирование GSK3B контролирует стабильность BMAL1 и предготавливает его для последующего убиквитинирования[25].
Другие функции
Ген Arntl у крыс расположен в локусах восприимчивости гипертензии 1 хромосомы. Изучение мононуклеотидных полиморфизмов (SNP) в этих локусах обнаружило два полиморфизма, которые происходили в последовательности, кодирующей Arntl, и были связаны с диабетом II типа и гипертонией. При переходе от модели крысы к человеческой модели это исследование предполагает причинную роль вариации гена Arntl в патологии диабета II типа[26]. Недавние данные о фенотипах также предполагают, что этот ген[27] и его партнёр clock[28] играют роль в регуляции гомеостаза и метаболизма глюкозы, и при нарушениях могут привести к гипоинсулинемии или диабету[29]. Что касается других функций, то в другом исследовании показано, как комплекс CLOCK:BMAL1 повышает активность промотора LDLR человека, показывая, что ген Arntl также играет роль в гомеостазе холестерина[30]. Кроме того, было обнаружено, что экспрессия гена Arntl, наряду с генами других генов клеточного ядра, была ниже у пациентов с биполярным расстройством, что указывает на проблему с циркадной функцией у этих пациентов[31]. Arntl, Npas2 и Per2 также были связаны с сезонным аффективным расстройством у людей[32]. Наконец, Arntl был идентифицирован посредством функционального генетического скрининга как предполагаемого регулятора пути онкосупрессора р53, потенциально возможное участие в циркадных ритмах, которые проявляются злокачественными клетками[33].
Взаимодействия с белками
ARNTL взаимодействует со следующими белками:
Примечания
- ARNTL aryl hydrocarbon receptor nuclear translocator-like [ Homo sapiens (human) ]. National Center for Biotechnology Information.
- UniProt, O00327 (англ.).
- Hogenesch J.B., Chan W.K., Jackiw V.H., Brown R.C., Gu Y.Z., Pray-Grant M., Perdew G.H., Bradfield C.A. Characterization of a subset of the basic-helix-loop-helix-PAS superfamily that interacts with components of the dioxin signaling pathway (англ.) // Journal of Biological Chemistry : journal. — 1997. — March (vol. 272, no. 13). — P. 8581—8593. — doi:10.1074/jbc.272.13.8581. — PMID 9079689.
- Ikeda M., Nomura M. cDNA cloning and tissue-specific expression of a novel basic helix-loop-helix/PAS protein (BMAL1) and identification of alternatively spliced variants with alternative translation initiation site usage (англ.) // Biochemical and Biophysical Research Communications : journal. — 1997. — April (vol. 233, no. 1). — P. 258—264. — doi:10.1006/bbrc.1997.6371. — PMID 9144434.
- Hogenesch J.B., Gu Y.Z., Jain S., Bradfield C.A. The basic-helix-loop-helix-PAS orphan MOP3 forms transcriptionally active complexes with circadian and hypoxia factors (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1998. — May (vol. 95, no. 10). — P. 5474—5479. — doi:10.1073/pnas.95.10.5474. — . — PMID 9576906.
- Jin X., Shearman L.P., Weaver D.R., Zylka M.J., de Vries G.J., Reppert S.M. A molecular mechanism regulating rhythmic output from the suprachiasmatic circadian clock (англ.) // Cell : journal. — Cell Press, 1999. — January (vol. 96, no. 1). — P. 57—68. — doi:10.1016/S0092-8674(00)80959-9. — PMID 9989497.
- Bunger M.K., Wilsbacher L.D., Moran S.M., Clendenin C., Radcliffe L.A., Hogenesch J.B., Simon M.C., Takahashi J.S., Bradfield C.A. Mop3 is an essential component of the master circadian pacemaker in mammals (англ.) // Cell : journal. — Cell Press, 2000. — December (vol. 103, no. 7). — P. 1009—1017. — doi:10.1016/S0092-8674(00)00205-1. — PMID 11163178.
- Huang N., Chelliah Y., Shan Y., Taylor C.A., Yoo S.H., Partch C., Green C.B., Zhang H., Takahashi J.S. Crystal structure of the heterodimeric CLOCK:BMAL1 transcriptional activator complex (англ.) // Science : journal. — 2012. — July (vol. 337, no. 6091). — P. 189—194. — doi:10.1126/science.1222804. — . — PMID 22653727.
- Wang Z., Wu Y., Li L., Su X.D. Intermolecular recognition revealed by the complex structure of human CLOCK-BMAL1 basic helix-loop-helix domains with E-box DNA (англ.) // Cell Research : journal. — 2013. — February (vol. 23, no. 2). — P. 213—224. — doi:10.1038/cr.2012.170. — PMID 23229515.
- Harfmann B.D., Schroder E.A., Esser K.A. Circadian rhythms, the molecular clock, and skeletal muscle (англ.) // Journal of Biological Rhythms : journal. — 2015. — April (vol. 30, no. 2). — P. 84—94. — doi:10.1177/0748730414561638. — PMID 25512305.
- Buhr E.D., Takahashi J.S. Molecular components of the Mammalian circadian clock (англ.) // Handbook of Experimental Pharmacology : journal. — 2013. — Vol. Handbook of Experimental Pharmacology, no. 217. — P. 3—27. — ISBN 978-3-642-25949-4. — doi:10.1007/978-3-642-25950-0_1. — PMID 23604473.
- Bollinger T., Schibler U. Circadian rhythms - from genes to physiology and disease (англ.) // Swiss Medical Weekly : journal. — 2014. — Vol. 144. — P. w13984. — doi:10.4414/smw.2014.13984. — PMID 25058693.
- Maywood E.S., Chesham J.E., Smyllie N.J., Hastings M.H. The Tau mutation of casein kinase 1ε sets the period of the mammalian pacemaker via regulation of Period1 or Period2 clock proteins (англ.) // Journal of Biological Rhythms : journal. — 2014. — April (vol. 29, no. 2). — P. 110—118. — doi:10.1177/0748730414520663. — PMID 24682205.
- Asher G., Gatfield D., Stratmann M., Reinke H., Dibner C., Kreppel F., Mostoslavsky R., Alt F.W., Schibler U. SIRT1 regulates circadian clock gene expression through PER2 deacetylation (англ.) // Cell : journal. — Cell Press, 2008. — July (vol. 134, no. 2). — P. 317—328. — doi:10.1016/j.cell.2008.06.050. — PMID 18662546.
- Ueda H.R., Chen W., Adachi A., Wakamatsu H., Hayashi S., Takasugi T., Nagano M., Nakahama K., Suzuki Y., Sugano S., Iino M., Shigeyoshi Y., Hashimoto S. A transcription factor response element for gene expression during circadian night (англ.) // Nature. — 2002. — August (vol. 418, no. 6897). — P. 534—539. — doi:10.1038/nature00906. — . — PMID 12152080.
- Akashi M., Takumi T. The orphan nuclear receptor RORalpha regulates circadian transcription of the mammalian core-clock Bmal1 (англ.) // Nature Structural & Molecular Biology : journal. — 2005. — May (vol. 12, no. 5). — P. 441—448. — doi:10.1038/nsmb925. — PMID 15821743.
- Guillaumond F., Dardente H., Giguère V., Cermakian N. Differential control of Bmal1 circadian transcription by REV-ERB and ROR nuclear receptors (англ.) // Journal of Biological Rhythms : journal. — 2005. — October (vol. 20, no. 5). — P. 391—403. — doi:10.1177/0748730405277232. — PMID 16267379.
- Ueda H.R., Hayashi S., Chen W., Sano M., Machida M., Shigeyoshi Y., Iino M., Hashimoto S. System-level identification of transcriptional circuits underlying mammalian circadian clocks (англ.) // Nature Genetics : journal. — 2005. — February (vol. 37, no. 2). — P. 187—192. — doi:10.1038/ng1504. — PMID 15665827.
- Liu A.C., Tran H.G., Zhang E.E., Priest A.A., Welsh D.K., Kay S.A. Redundant function of REV-ERBα and β and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms (англ.) // PLOS Genetics : journal / Takahashi, Joseph S.. — 2008. — February (vol. 4, no. 2). — P. e1000023. — doi:10.1371/journal.pgen.1000023. — PMID 18454201.
- Hirayama J., Sahar S., Grimaldi B., Tamaru T., Takamatsu K., Nakahata Y., Sassone-Corsi P. CLOCK-mediated acetylation of BMAL1 controls circadian function (англ.) // Nature : journal. — 2007. — December (vol. 450, no. 7172). — P. 1086—1090. — doi:10.1038/nature06394. — . — PMID 18075593.
- Lee J., Lee Y., Lee M.J., Park E., Kang S.H., Chung C.H., Lee K.H., Kim K. Dual modification of BMAL1 by SUMO2/3 and ubiquitin promotes circadian activation of the CLOCK/BMAL1 complex (англ.) // Molecular and Cellular Biology : journal. — 2008. — October (vol. 28, no. 19). — P. 6056—6065. — doi:10.1128/MCB.00583-08. — PMID 18644859.
- Eide E.J., Vielhaber E.L., Hinz W.A., Virshup D.M. The circadian regulatory proteins BMAL1 and cryptochromes are substrates of casein kinase Iepsilon (англ.) // Journal of Biological Chemistry : journal. — 2002. — May (vol. 277, no. 19). — P. 17248—17254. — doi:10.1074/jbc.m111466200. — PMID 11875063.
- Sanada K., Okano T., Fukada Y. Mitogen-activated protein kinase phosphorylates and negatively regulates basic helix-loop-helix-PAS transcription factor BMAL1 (англ.) // Journal of Biological Chemistry : journal. — 2002. — January (vol. 277, no. 1). — P. 267—271. — doi:10.1074/jbc.m107850200. — PMID 11687575.
- Tamaru T., Hirayama J., Isojima Y., Nagai K., Norioka S., Takamatsu K., Sassone-Corsi P. CK2alpha phosphorylates BMAL1 to regulate the mammalian clock (англ.) // Nature Structural & Molecular Biology : journal. — 2009. — April (vol. 16, no. 4). — P. 446—448. — doi:10.1038/nsmb.1578. — PMID 19330005.
- Sahar S., Zocchi L., Kinoshita C., Borrelli E., Sassone-Corsi P. Regulation of BMAL1 protein stability and circadian function by GSK3beta-mediated phosphorylation (англ.) // PLoS ONE : journal. — 2010. — January (vol. 5, no. 1). — P. e8561. — doi:10.1371/journal.pone.0008561. — . — PMID 20049328.
- Woon P.Y., Kaisaki P.J., Bragança J., Bihoreau M.T., Levy J.C., Farrall M., Gauguier D. Aryl hydrocarbon receptor nuclear translocator-like (BMAL1) is associated with susceptibility to hypertension and type 2 diabetes (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2007. — September (vol. 104, no. 36). — P. 14412—14417. — doi:10.1073/pnas.0703247104. — . — PMID 17728404.
- Rudic R.D., McNamara P., Curtis A.M., Boston R.C., Panda S., Hogenesch J.B., Fitzgerald G.A. BMAL1 and CLOCK, two essential components of the circadian clock, are involved in glucose homeostasis (англ.) // PLOS Biology : journal. — 2004. — November (vol. 2, no. 11). — P. e377. — doi:10.1371/journal.pbio.0020377. — PMID 15523558.
- Turek F.W., Joshu C., Kohsaka A., Lin E., Ivanova G., McDearmon E., Laposky A., Losee-Olson S., Easton A., Jensen D.R., Eckel R.H., Takahashi J.S., Bass J. Obesity and metabolic syndrome in circadian Clock mutant mice (англ.) // Science : journal. — 2005. — May (vol. 308, no. 5724). — P. 1043—1045. — doi:10.1126/science.1108750. — . — PMID 15845877.
- Marcheva B., Ramsey K.M., Buhr E.D., Kobayashi Y., Su H., Ko C.H., Ivanova G., Omura C., Mo S., Vitaterna M.H., Lopez J.P., Philipson L.H., Bradfield C.A., Crosby S.D., JeBailey L., Wang X., Takahashi J.S., Bass J. Disruption of the clock components CLOCK and BMAL1 leads to hypoinsulinaemia and diabetes (англ.) // Nature : journal. — 2010. — July (vol. 466, no. 7306). — P. 627—631. — doi:10.1038/nature09253. — . — PMID 20562852.
- Lee Y.J., Han D.H., Pak Y.K., Cho S.H. Circadian regulation of low density lipoprotein receptor promoter activity by CLOCK/BMAL1, Hes1 and Hes6 (англ.) // Experimental and Molecular Medicine : journal. — 2012. — November (vol. 44, no. 11). — P. 642—652. — doi:10.3858/emm.2012.44.11.073. — PMID 22913986.
- Yang S., Van Dongen H.P., Wang K., Berrettini W., Bućan M. Assessment of circadian function in fibroblasts of patients with bipolar disorder (англ.) // Molecular Psychiatry : journal. — 2009. — February (vol. 14, no. 2). — P. 143—155. — doi:10.1038/mp.2008.10. — PMID 18301395.
- Partonen T., Treutlein J., Alpman A., Frank J., Johansson C., Depner M., Aron L., Rietschel M., Wellek S., Soronen P., Paunio T., Koch A., Chen P., Lathrop M., Adolfsson R., Persson M.L., Kasper S., Schalling M., Peltonen L., Schumann G. Three circadian clock genes Per2, Arntl, and Npas2 contribute to winter depression (англ.) // Annals of Medicine : journal. — 2007. — Vol. 39, no. 3. — P. 229—238. — doi:10.1080/07853890701278795. — PMID 17457720.
- Mullenders J., Fabius A.W., Madiredjo M., Bernards R., Beijersbergen R.L. A large scale shRNA barcode screen identifies the circadian clock component ARNTL as putative regulator of the p53 tumor suppressor pathway (англ.) // PLoS ONE : journal. — 2009. — Vol. 4, no. 3. — P. e4798. — doi:10.1371/journal.pone.0004798. — . — PMID 19277210.
- Ooe N., Saito K., Mikami N., Nakatuka I., Kaneko H. Identification of a novel basic helix-loop-helix-PAS factor, NXF, reveals a Sim2 competitive, positive regulatory role in dendritic-cytoskeleton modulator drebrin gene expression (англ.) // Molecular and Cellular Biology : journal. — 2004. — January (vol. 24, no. 2). — P. 608—616. — doi:10.1128/MCB.24.2.608-616.2004. — PMID 14701734.
- McNamara P., Seo S.B., Rudic R.D., Sehgal A., Chakravarti D., FitzGerald G.A. Regulation of CLOCK and MOP4 by nuclear hormone receptors in the vasculature: a humoral mechanism to reset a peripheral clock (англ.) // Cell : journal. — Cell Press, 2001. — June (vol. 105, no. 7). — P. 877—889. — doi:10.1016/S0092-8674(01)00401-9. — PMID 11439184.
- Takahata S., Ozaki T., Mimura J., Kikuchi Y., Sogawa K., Fujii-Kuriyama Y. Transactivation mechanisms of mouse clock transcription factors, mClock and mArnt3 (англ.) // Genes to Cells : journal. — 2000. — September (vol. 5, no. 9). — P. 739—747. — doi:10.1046/j.1365-2443.2000.00363.x. — PMID 10971655.
- Xu H., Gustafson C.L., Sammons P.J., Khan S.K., Parsley N.C., Ramanathan C., Lee H.W., Liu A.C., Partch C.L. Cryptochrome 1 regulates the circadian clock through dynamic interactions with the BMAL1 C terminus (англ.) // Nature Structural & Molecular Biology : journal. — 2015. — June (vol. 22, no. 6). — P. 476—484. — doi:10.1038/nsmb.3018. — PMID 25961797.