Ensembl
Ensembl — совместный научный проект Европейского института биоинформатики и Института Сенгера. Основной задачей этого проекта является обеспечение специалистов интегрированным доступом к базам данных, касающихся строения геномов более 50 видов позвоночных, включая человека (Homo sapiens), мышь (Mus musculus), крысу (Rattus norvegicus), рыбку Данио-рерио (Danio rerio) и др.[1]. Проект был запущен в 1999 году перед завершением проекта «Геном человека»[2].

Базы данных Ensembl регулярно обновляются с частотой не менее двух раз в год. Текущая версия проекта 88 была опубликована 29 марта 2017 года[3]. Свежие новости проекта публикуются на официальном веб-сайте. Там же можно найти информацию об очных образовательных мероприятиях по работе с Ensembl[4]. Основам работы с системой можно также научиться, просмотрев тематические видео на сайте Ensembl и EMBL-EBI.
Аннотация генома
Основные элементы генома позвоночных
Проект Ensembl сосредоточен на предоставлении подробной информации о геномах позвоночных животных. Типичный размер такого генома составляет миллиарды пар оснований. Например, геном мыши (Mus musculus) и геном человека (Homo sapiens) содержат около 3 миллиардов пар оснований. Только несколько процентов генома представляют собой кодирующие последовательности, соответствующие примерно 20—25 тысячам генов в случае человека[5]. Кодирующие последовательности обладают неслучайной структурой, что позволяет обнаружить их при анализе генома. Некодирующие последовательности генома в некоторых случаях также являются биологически функциональными, подразделяясь на псевдогены, гены транспортной и рибосомной РНК, гены длинных некодирующих РНК, малых ядерных РНК, малых ядрышковых РНК, микроРНК и т.д. Работа с элементами генома возможна только при наличии информации о положении этого элемента и взаимодействии с другими. Разметка положения каждого такого элемента называется аннотацией генома[6].
Аннотация генома может быть проведена как вручную коллективом экспертов, так и с использованием автоматических программных подходов, как это реализовано в Ensembl[7].
Система аннотации Ensembl
Стандартная процедура аннотации Ensembl занимает до 4 месяцев и состоит из нескольких стадий[8]. Вначале проводится автоматическая маскировка повторов и предсказание положения генов. Затем на геном выравниваются известные кодирующие белки последовательности данного организма, полученные экспериментально. Если для участка генома такой последовательности нет, в ходе последующей стадии для этой цели используются последовательности близких видов. Помимо этого на геном наносится информация об известных видоспецифичных последовательностях кДНК и EST. Когда возможно, на геном также накладываются данные экспериментов по РНК-секвенированию[9].
Для геномов человека и мыши стандартный процесс аннотации дополняется аннотацией проекта HAVANA. Объединенная аннотация Ensembl/HAVANA составляет набор генов человека и мыши GENCODE[10].
В номенклатуре Ensembl гену может быть присвоено 3 статуса: known, novel, merged. Статус known указывает на то, что данный участок соотносится с известной последовательностью этого организма из публичных баз данных UniProtKB и NCBI RefSeq. В случае, если есть совпадение только с последовательностью другого организма, гену присваивается статус novel. Статус merged указывает на полное совпадение аннотаций Ensembl и HAVANA[8].
Геномный браузер Ensembl
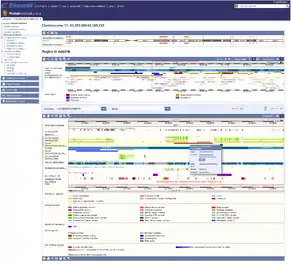
Первичная цель проекта Ensembl заключается в автоматическом анализе и аннотировании геномов позвоночных, а также предоставлении доступа к этим геномам. Геномный браузер Ensembl способен визуализировать имеющиеся в базе Ensembl геномы и их аннотации в различном масштабе, от целого кариотипа до конкретного участка последовательности генома в текстовом виде. Аннотированные элементы отображаются в виде полос (треков) относительно референсного генома. Визуализация треков может быть настроена пользователем под собственные нужды. Дополнительная информация по каждому элементу аннотации доступна во всплывающих окнах при наведении курсора на элемент. Пользователям доступна возможность загружать и визуализировать собственные данные по геномной аннотации. Сделать это можно либо используя сервер DAS (Distributed Annotation System), либо загрузив файл в поддерживаемом формате (BigBED, BigWig, VCF, BAM и другие)[11][12][13][14].
API и другие способы доступа
Для хранения информации Ensembl использует реляционные базы данных на MySQL. Для получения информации из баз Ensembl используется набор API (интерфейс программирования приложений), написанных на Perl. API позволяют не зависеть от изменений в структуре базы данных сторонним приложениям. API Ensembl используется в веб-интерфейсе проекта для представления данных, а также может быть загружен пользователем и применяться для написания скриптов для автоматизации получения данных из баз Ensembl. Информация по загрузке, установке и использованию API Ensembl содержится на сайте проекта[15].
API Ensembl подразделяется на секции по типам обрабатываемых данных: Ensembl Core API (для работы с генами, последовательностями и другими элементами автоматической аннотации), Ensembl-Compara API (для работы с данными по сравнительной геномике), Ensembl-Variation API (для работы с данными о однонуклеотидных полиморфизмах, соматических мутациях, структурных вариациях), Ensembl-Regulation API (для работы с данными по регуляции генома) и другие[16].
Для доступа к базе Ensembl с использованием клиента на другом языке программирования используется сервер Ensembl REST[17]. Для получения больших объемов данных может использоваться сервис BioMart. Кроме того для скачивания полных баз Ensembl на MySQL можно использовать FTP-сервер проекта.
Доступные инструменты
На сайте Ensembl доступен ряд инструментов для обработки данных как из базы Ensembl, так и загруженных пользователем[18]. Для поиска заданной последовательности по всем геномам Ensembl используются алгоритмы BLAT или BLAST. Присутствует инструмент для загрузки данных из базы Ensembl в видоизменённом формате (File Chameleon), а также для перевода формата данных между разными сборками геномов и релизами Ensembl.
Variant effect predictor
Ensembl Variant effect predictor (VEP) — это инструмент для анализа и аннотации геномных вариаций в кодирующих и некодирующих участках. VEP аннотирует геномные вариации, опираясь на широкую выборку данных базы Ensembl, включая транскрипты, регуляторные области, частоты ранее наблюдавшихся вариаций, клинические данные и предсказания биофизических последствий вариаций. Возможен анализ двух категорий вариаций: малые точно определенные вариации (инсерции, делеции, тандемные повторы, однонуклеотидные полиморфизмы) либо более крупные структурные вариации генома (изменения копийности генов, крупные инсерции или делеции). VEP доступен в виде сервиса на веб-сайте Ensembl, в виде отдельного Perl-скрипта, а также через Ensembl REST[19].
Партнёрские проекты
Ensembl Genomes
Изначально проект Ensembl специализировался на геномах позвоночных животных, однако увеличение количества информации о геномах других живых существ привело к появлению в 2009 году проекта Ensembl Genomes, использующего платформу, инструменты и систему аннотации Ensembl[20]. В рамках данного проекта было создано 5 подразделений:
- Ensembl Bacteria предоставляет доступ к более чем 40 тысячам аннотированных бактериальных геномов
- Ensembl Fungi содержит 569 геномов грибов
- Ensembl Plants содержит 44 генома растений
- Ensembl Protists содержит 150 геномов протистов
- Ensembl Metazoa содержит 65 геномов беспозвоночных животных
Сборка геномов Ensembl Genomes выходит одновременно для всех подразделений и независимо от основного проекта. Текущая версия сервиса 34, последнее обновление было совершено в декабре 2016 года[21].
Ensembl Pre!
Доступ к геномам, находящимся в процессе аннотации, осуществляется с помощью сервиса Ensembl Pre!. По состоянию на 2017 год доступна информация о геномах 17 организмов. Последнее обновление сервиса было проведено 19 января 2015 года[22].
Примечания
- Paul Flicek, Bronwen L. Aken, Benoit Ballester, Kathryn Beal, Eugene Bragin. Ensembl's 10th year (англ.) // Nucleic Acids Research. — 2010-01-01. — Vol. 38, iss. suppl_1. — P. D557–D562. — ISSN 0305-1048. — doi:10.1093/nar/gkp972.
- Paul Flicek, M. Ridwan Amode, Daniel Barrell, Kathryn Beal, Simon Brent. Ensembl 2011 (англ.) // Nucleic Acids Research. — 2011-01-01. — Vol. 39, iss. suppl_1. — P. D800–D806. — ISSN 0305-1048. — doi:10.1093/nar/gkq1064.
- Ensembl 88 has been released! (29 марта 2017).
- Ensembl workshops. Ensembl.
- Human assembly and gene annotation. Ensembl (March 2017).
- Roger P. Alexander, Gang Fang, Joel Rozowsky, Michael Snyder, Mark B. Gerstein. Annotating non-coding regions of the genome (англ.) // Nature Reviews Genetics. — Vol. 11, iss. 8. — P. 559–571. — doi:10.1038/nrg2814.
- Val Curwen, Eduardo Eyras, T. Daniel Andrews, Laura Clarke, Emmanuel Mongin. The Ensembl Automatic Gene Annotation System (англ.) // Genome Research. — 2004-05-01. — Vol. 14, iss. 5. — P. 942–950. — doi:10.1101/gr.1858004.
- Ensembl annotation (недоступная ссылка). Дата обращения: 14 апреля 2017. Архивировано 15 апреля 2017 года.
- Bronwen L. Aken, Sarah Ayling, Daniel Barrell, Laura Clarke, Valery Curwen. The Ensembl gene annotation system (англ.) // Database. — 2016-01-01. — Vol. 2016. — doi:10.1093/database/baw093.
- Why do human and mouse gene counts change between GENCODE releases? (недоступная ссылка). GencodeGenes (13 сентября 2016). Дата обращения: 15 апреля 2017. Архивировано 24 мая 2017 года.
- Andrew Yates, Wasiu Akanni, M. Ridwan Amode, Daniel Barrell, Konstantinos Billis. Ensembl 2016 (англ.) // Nucleic Acids Research. — 2016-01-04. — Vol. 44, iss. D1. — P. D710–D716. — ISSN 0305-1048. — doi:10.1093/nar/gkv1157.
- Giulietta M. Spudich, Xosé M. Fernández-Suárez. Touring Ensembl: A practical guide to genome browsing (англ.) // BMC Genomics. — 2010-01-01. — Vol. 11. — P. 295. — ISSN 1471-2164. — doi:10.1186/1471-2164-11-295.
- Giulietta Spudich, Xosé M. Fernández-Suárez, Ewan Birney. Genome browsing with Ensembl: a practical overview (англ.) // Briefings in Functional Genomics. — 2007-09-01. — Vol. 6, iss. 3. — P. 202–219. — ISSN 2041-2649. — doi:10.1093/bfgp/elm025.
- Xosé M. Fernández-Suárez, Michael K. Schuster. Using the Ensembl Genome Server to Browse Genomic Sequence Data (англ.) // Current Protocols in Bioinformatics. — John Wiley & Sons, Inc., 2002-01-01. — ISBN 9780471250951. — doi:10.1002/0471250953.bi0115s30.
- Arne Stabenau, Graham McVicker, Craig Melsopp, Glenn Proctor, Michele Clamp. The Ensembl Core Software Libraries (англ.) // Genome Research. — 2004-05-01. — Vol. 14, iss. 5. — P. 929–933. — doi:10.1101/gr.1857204.
- Doxygen Perl documentation (англ.). www.ensembl.org. Дата обращения: 14 апреля 2017.
- Andrew Yates, Kathryn Beal, Stephen Keenan, William McLaren, Miguel Pignatelli. The Ensembl REST API: Ensembl Data for Any Language (англ.) // Bioinformatics. — 2015-01-01. — Vol. 31, iss. 1. — P. 143–145. — ISSN 1367-4803. — doi:10.1093/bioinformatics/btu613.
- Ensembl Tools (англ.). www.ensembl.org. Дата обращения: 14 апреля 2017.
- William McLaren, Laurent Gil, Sarah E. Hunt, Harpreet Singh Riat, Graham R. S. Ritchie. The Ensembl Variant Effect Predictor (англ.) // Genome Biology. — 2016-01-01. — Vol. 17. — P. 122. — ISSN 1474-760X. — doi:10.1186/s13059-016-0974-4.
- Paul Julian Kersey, James E. Allen, Irina Armean, Sanjay Boddu, Bruce J. Bolt. Ensembl Genomes 2016: more genomes, more complexity (англ.) // Nucleic Acids Research. — 2016-01-04. — Vol. 44, iss. D1. — P. D574–D580. — ISSN 0305-1048. — doi:10.1093/nar/gkv1209.
- Ensembl Genomes.
- Murphy Dan (Genebuild). New Ensembl Pre! sites. Ensembl Blog. Ensembl (19 января 2015).
