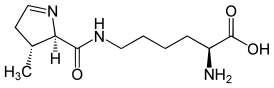
Пирролизин
Пирролизин — это природная аминокислота, которая включается во время трансляции в полипептидные цепи некоторых белков, кодируемых генами некоторых метаногенных архей, у человека она отсутсвует. Он содержит α-аминогруппу (которая находится в протонированной форме NH3+ в биологических условиях), группу карбоновых кислот (которая находится в депротонированной форме COO- в биологических условиях). Его пирролиновая боковая цепь сходна с цепью лизина по своей основности и положительному заряду при нейтральном рН. Пирролизин входит в состав некоторых ферментов метаболизма метана.
| Пирролизин | |
|---|---|
  | |
| Общие | |
| Хим. формула | C12H21N3O3 |
| Физические свойства | |
| Молярная масса | 255,313 г/моль |
| Классификация | |
| Рег. номер CAS | 448235-52-7 |
| PubChem | 5460671 |
| SMILES | |
| InChI | |
| ChEBI | 21860 |
| ChemSpider | 4574156 |
| Приведены данные для стандартных условий (25 °C, 100 кПа), если не указано иное. | |
Для обозначения пирролизина ИЮПАК рекомендует трёхбуквенное сокращение Pyl и однобуквенное O.
Генетика
Почти все гены переводятся с использованием только 20 стандартных аминокислотных строительных блоков. Две необычные генетически закодированные аминокислоты - селеноцистеин и пирролизин. Пирролизин был обнаружен в 2002 году на активном участке фермента метилтрансферазы из метанопродуцирующего археона, Methanosarcina barkeri[1][2]. Эта аминокислота кодируется UAG (обычно стоп-кодон), а ее синтез и включение в белок опосредуется биологическим механизмом, кодируемым кластером генов pylTSBCD[3].
Строение
Как определено с помощью рентгеновской кристаллографии[2] и масс-спектрометрии, пирролизин представляет собой лизин с ϵ-азотом в амидной связи с (4r,5r)-4-замещенным пирролин-5-карбоксилатом[4].
Синтез
Пирролизин синтезируется в естественной среде путем соединения двух молекул L-лизина. Одна молекула лизина сначала преобразуется в (R)-3-метил-D-орнитин, который затем лигируется со вторым лизином. Группа NH2 удаляется, после чего следует стадия циклизации и дегидратации с получением L-пирролизина[5].
Генетическое кодирование
Пирролизин является производным аминокислоты лизина и кодируется кодоном UAG. Обычно этот кодон выполняет роль стоп-кодона. Он используется реже, чем другие стоп-кодоны, и, если встречается в открытой рамке считывания, за ним, как правило, следуют другие стоп-кодоны.
Рядом с кластером генов переноса метильной группы археи Methanosarcina barkeri располагается ген pylT, который кодирует необычную тРНК с антикодоном CUA. Соседний ген pylS кодирует аминоацил-тРНК-синтетазу второго класса, которая присоединяет пирролизин к тРНК-продукту гена pylT. Оперон, содержащий гены pylT и pylS также найден в геномах других секвенированных представителей семейства Methanosarcinaceae. Гомологи генов pylS и pylT обнаружены также в грам-положительной бактерии Desulfitobacterium hafniense, хотя функции этих гомологов у данной бактерии неизвестны.[6]
Первоначально было показано, что продукт гена pylT — тРНК с антикодоном (CUA) — может быть «заряжена» аминокислотой лизином с помощью белка PylS. Относительно недавно[когда?] было показано, что тРНК с антикодоном CUA может быть «заряжена» лизином в условиях in vitro последовательным взаимодействием с лизиновыми тРНК-синтетазами первого и второго класса из M. barkeri. Последние данные указывают на то, что происходит прямое присоединение пирролизина к тРНК с антикодоном CUA при помощи белкового продукта гена pylS. Это означает, что пирролизин представляет собой 22-ю аминокислоту, закодированную генетически.[7]
Каталитическая функция
Дополнительное пирролиновое кольцо включено в активный центр нескольких метилтрансфераз, где, как полагают, оно вращается относительно свободно. Считается, что кольцо участвует в позиционировании и отображении метильной группы метиламина для атаки корриноидным кофактором. Предложенная модель состоит в том, что соседний остаток , несущий карбоновую кислоту , глутамат, протонируется, и протон затем может быть перенесен в азот иминового кольца, подвергая соседний кольцевой углерод нуклеофильному присоединению метиламином. Положительно заряженный азот, созданный этим взаимодействием, может затем взаимодействовать с депротонированным глутаматом, вызывая сдвиг в ориентации кольца и подвергая метильную группу, полученную из метиламина, связывающей расщелине, где он может взаимодействовать с корриноидом. Таким образом, чистый CH3+ переносится на атом кобальта кофактора с изменением состояния окисления с I на III. Затем высвобождается аммиак метиламинового происхождения, восстанавливая исходный имин[2].
См. также
- Генетический код
- Трансляция
- Селеноцистеин, 21-я генетически закодированная аминокислота
Примечания
- G. Srinivasan. Pyrrolysine Encoded by UAG in Archaea: Charging of a UAG-Decoding Specialized tRNA // Science. — 2002-05-24. — Т. 296, вып. 5572. — С. 1459–1462. — doi:10.1126/science.1069588.
- B. Hao. A New UAG-Encoded Residue in the Structure of a Methanogen Methyltransferase // Science. — 2002-05-24. — Т. 296, вып. 5572. — С. 1462–1466. — doi:10.1126/science.1069556.
- Michael Rother, Joseph A. Krzycki. Selenocysteine, Pyrrolysine, and the Unique Energy Metabolism of Methanogenic Archaea (англ.) // Archaea. — 2010. — Vol. 2010. — P. 1–14. — ISSN 1472-3654 1472-3646, 1472-3654. — doi:10.1155/2010/453642.
- Jitesh A. Soares, Liwen Zhang, Rhonda L. Pitsch, Nanette M. Kleinholz, R. Benjamin Jones. The Residue Mass of L-Pyrrolysine in Three Distinct Methylamine Methyltransferases // Journal of Biological Chemistry. — 2005-11. — Т. 280, вып. 44. — С. 36962–36969. — ISSN 0021-9258. — doi:10.1074/jbc.m506402200.
- Marsha A. Gaston, Liwen Zhang, Kari B. Green-Church, Joseph A. Krzycki. The complete biosynthesis of the genetically encoded amino acid pyrrolysine from lysine (англ.) // Nature. — 2011-03. — Vol. 471, iss. 7340. — P. 647–650. — ISSN 1476-4687 0028-0836, 1476-4687. — doi:10.1038/nature09918.
- John F. Atkins and Ray Gesteland. The 22nd Amino Acid (англ.) // Science. — 2002. — Vol. 296, no. 5572. — P. 1409—1410. — doi:10.1126/science.1073339. — PMID 12029118.
- J. Krzycki. The direct genetic encoding of pyrrolysine (неопр.) // Curr Opin Microbiol. — 2005. — Т. 8, № 6. — С. 706—712. — doi:10.1016/j.mib.2005.10.009. — PMID 16256420.