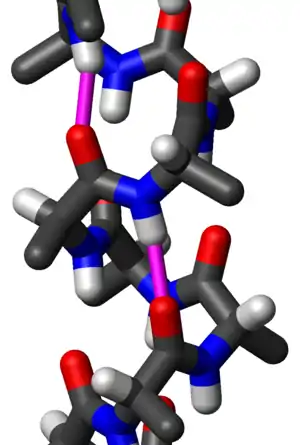
Пи-спираль
Пи-спираль (или π-спираль) — это тип вторичной структуры, обнаруженной в белках[1]. Открытые кристаллографом Барбарой Лоу в 1952 году[2] и когда-то считавшиеся редкостью, короткие π-спирали обнаруживаются в 15 % известных белковых структур и считаются эволюционной адаптацией, полученной путем вставки одной аминокислоты в α-спираль[3]. Поскольку такие вставки сильно дестабилизируют белковую цепь[4] образование π-спиралей будет иметь тенденцию быть подверженными эволюционному отбору, если только это не обеспечит некоторые функциональные преимущества для белка. Следовательно, π-спирали обычно находятся рядом с функциональными сайтами белков[3][5][6].
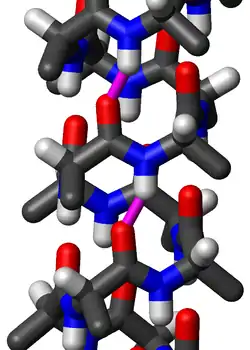
Стандартная структура
Аминокислоты в стандартной π-спирали расположены в виде правозакрученной спиральной структуры. Каждая аминокислота соответствует повороту спирали на 87° (то есть спираль имеет 4,1 остатка на оборот) и сдвигу на 1,15 Å (0,115 нм) вдоль оси спирали. Наиболее важно то, что группа NH аминокислоты образует водородную связь с группой C = O аминокислоты пятью остатками ранее; эта повторяющаяся i + 5 → i водородная связь определяет π-спираль. Подобные структуры построения встречаются у спирали 310 (i + 3 → i водородная связь) и α-спирали (i + 4 → i водородная связь).
Большинство π-спиралей имеют длину всего 7 остатков и не имеют регулярно повторяющихся (φ , ψ) двугранных углов по всей структуре, как у α-спиралей или β-листов. Однако можно сделать некоторые обобщения. Когда первая и последняя пары аминокислотных остатков исключены, двугранные углы существуют таким образом, что ψ двугранный угол одного остатка и φ двугранный угол следующего остатка составляют примерно −125°. Сумма первой и последней пары остатков равна −95° и −105° соответственно. Для сравнения, сумма двугранных углов для спирали 310 составляет примерно −75°, тогда как для α-спирали примерно −105°. Пролин часто наблюдается сразу после окончания π-спиралей. Общая формула для угла поворота Ω на остаток любой полипептидной спирали с транс- изомерами дается уравнением
Левозакрученная структура
В принципе, левозакрученная версия π-спирали возможна, путем изменения знака (φ , ψ) двугранных углов на (55°, 70°). Эта псевдо- «зеркальная» спираль имеет примерно такое же количество остатков на виток (4.1) и шаг спирали (1.5 Å). Это ненастоящее зеркальное отображение, потому что аминокислотные остатки по-прежнему имеют левозакрученную хиральность. Длинная левозакрученная π-спираль вряд ли будет наблюдаться в белках, потому что среди встречающихся в природе аминокислот только глицин, вероятно, будет иметь положительные двугранные углы φ, такие как 55°.
π-спирали в природе
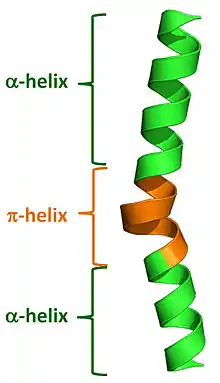
Обычно используемые программы автоматического определения вторичной структуры, такие как DSSP, предполагают, что <1 % белков содержат π-спираль. Эта неправильная характеристика является результатом того факта, что встречающиеся в природе π-спирали обычно имеют короткую длину (от 7 до 10 остатков) и почти всегда связаны с α-спиралями (то есть фланкированы) на обоих концах. Таким образом, почти все π-спирали скрыты в том смысле, что π-спиральные остатки неправильно отнесены либо к α-спирали, либо к «поворотам». Недавно разработанные программы были написаны для правильного аннотирования π-спиралей в белковых структурах, и они обнаружили, что каждый шестой белок (около 15 %) действительно содержит по крайней мере один π-спиральный сегмент[3].
Естественные π-спирали можно легко идентифицировать в структуре как «выпуклость» внутри более длинной α-спирали. Такие спиральные выпуклости ранее назывались α-аневризмами, α-выпуклостями, π-выпуклостями, широкими поворотами, петлевыми выходами и π-поворотами, но на самом деле являются π-спиралями, определяемыми их повторяющимися водородными связями i + 5 → i[3]. Данные свидетельствуют о том, что эти выпуклости или π-спирали создаются вставкой одной дополнительной аминокислоты в уже существующую α-спираль. Таким образом, α-спирали и π-спирали могут быть взаимно преобразованы путем вставки и удаления одной аминокислоты[4]. Учитывая как относительно высокую частоту появления π-спиралей, так и их отмеченную ассоциацию с функциональными сайтами (то есть активными центрами) белков, эта способность взаимного преобразования между α-спиралями и π-спиралями была важным механизмом изменения и диверсификации функциональности белков в ходе эволюции.
Одной из наиболее заметных групп белков, на функциональную диверсификацию которых, по-видимому, сильно повлиял такой эволюционный механизм, является ферритинподобное суперсемейство, которое включает ферритины, бактериоферритины, рубреритрины, рибонуклеотидредуктазы класса I и растворимые монооксигеназы метана. Растворимая метанмонооксигеназа является текущим рекордсменом по наибольшему количеству π-спиралей в одном ферменте (13 шт.). (код PDB 1MTY). Однако бактериальный гомолог Na+/Cl- зависимого транспортера нейротрансмиттеров (код PDB 2A65) является рекордсменом по количеству π-спиралей в одной пептидной цепи (8 шт.)[3].
См. также
Примечания
- “The Structure of Proteins: Two Hydrogen-Bonded Helical Configurations of the Polypeptide Chain”. Proc. Natl. Acad. Sci. U.S.A. 37 (4): 205—211. 1951. DOI:10.1073/pnas.37.4.205. PMID 14816373.
- (IUCr) Barbara Wharton Low (1920-2019). www.iucr.org. Дата обращения: 2 октября 2019.
- Richard B. Cooley, Daniel J. Arp, P. Andrew Karplus. Evolutionary Origin of a Secondary Structure: π-Helices as Cryptic but Widespread Insertional Variations of α-Helices That Enhance Protein Functionality (англ.) // Journal of Molecular Biology. — 2010-11. — Vol. 404, iss. 2. — P. 232–246. — doi:10.1016/j.jmb.2010.09.034.
- L. J. Keefe, J. Sondek, D. Shortle, E. E. Lattman. The alpha aneurism: a structural motif revealed in an insertion mutant of staphylococcal nuclease. (англ.) // Proceedings of the National Academy of Sciences. — 1993-04-15. — Vol. 90, iss. 8. — P. 3275–3279. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.90.8.3275.
- Weaver TM (2000). “The pi-helix translates structure into function”. Protein Science. 9 (1): 201—206. DOI:10.1110/ps.9.1.201. PMID 10739264.
- “Occurrence, conformational features and amino acid propensities for the pi-helix”. Protein Eng. 15 (5): 353—358. 2002. DOI:10.1093/protein/15.5.353. PMID 12034854.