Контроль качества мРНК
Контро́ль ка́чества мРНК[1] (англ. mRNA surveillance) — совокупность молекулярных механизмов, обеспечивающих отбраковывание дефектных мРНК и не допускающих их трансляцию[2][3]. Механизмы контроля качества мРНК действуют на разных этапах биогенеза мРНК. Как правило, они приводят к тому, что дефектные мРНК оказываются помеченными специфическим образом и благодаря этому распознаются ферментами-нуклеазами, разрушающими их[4].
Механизмы контроля качества мРНК описаны у бактерий и эукариот, причём у последних они протекают как в ядре, так и в цитоплазме[5]. Результатом работы этих механизмов в ядре является разрушение дефектных транскриптов, не допускающее их перемещение в цитоплазму. В цитоплазме транскрипты проверяются на наличие преждевременных стоп-кодонов[4][5].
В клетках эукариот описаны три механизма контроля качества мРНК: нонсенс-опосредованный распад (англ. nonsense-mediated decay, NMD), nonstop-деградация и no-go-деградация[6].
Нонсенс-опосредованный распад
Нонсенс-опосредованный распад (NMD) направлен на выявление и разрушение мРНК, содержащих преждевременные стоп-кодоны. Преждевременные стоп-кодоны могут появляться из-за мутаций в клетках зародышевой линии и соматических клетках, ошибках в транскрипции или посттранскрипционном процессинге мРНК[7][8]. Если такие мРНК не будут разрушаться, с них будут синтезированы усечённые белки, которые могут быть вредоносными для клетки[9]. Преждевременные стоп-кодоны задействованы в развитии примерно 30 % наследственных заболеваний. Таким образом, NMD играет важную роль в жизнедеятельности организма[10][11].
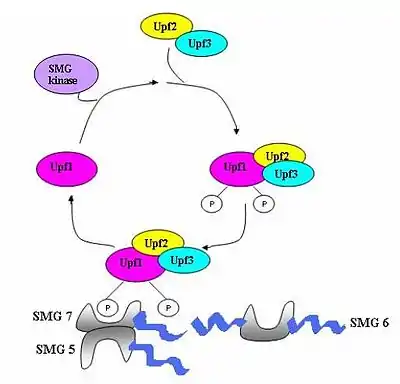
У дрожжей Saccharomyces cerevisiae и нематоды Caenorhabditis elegans в качестве необходимых транс-действующих факторов NMD функционируют три белка smg (smg1—7) и три белка UPF (Upf1—3)[12][13]. Соответствующие гены имеются и у плодовой мушки Drosophila melanogaster, и у млекопитающих, и их белковые продукты также участвуют в NMD[14]. В целом, у всех эукариот имеются белковые комплексы, задействованные в NMD: UPF1/SMG-2, UPF2/SMG-3 и UPF3/SMG-4. Однако их роли в NMD являются предметов споров. Неясно также, какие именно взаимодействия между этими белками происходят в действительности[15][12][14][16][17].
Было показано, что некоторые мРНК, содержащие преждевременные стоп-кодоны, не подвергаются NMD[18][19]. Как правило, в таких мРНК преждевременный стоп-кодон располагается в самом начале открытой рамки считывания[20]. Например, мРНК β-глобулина содержит преждевременный стоп-кодон в самом начале первого экзона и при этом не подвергается NMD. Детали механизма, позволяющего таким мРНК избежать разрушения, неизвестны. Было высказано предположение, что в нём задействован поли(А)-связывающий белок (PABP)[21].
У млекопитающих
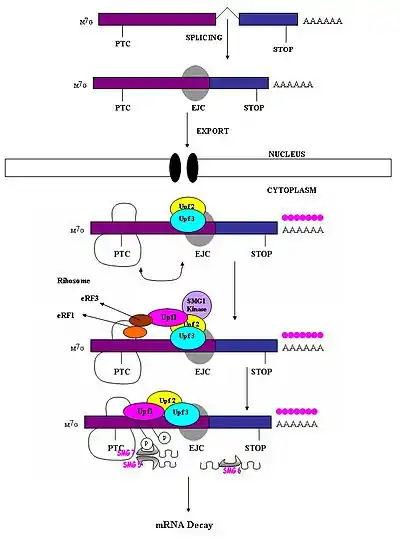
Было показано, что у млекопитающих для запуска разрушения мРНК важны нуклеотиды, расположенные на 50—54 и более нуклеотидов выше последнего места соединения двух экзонов[3][5][7][8][9][18]. Нуклеотиды, расположенные ниже этой точки, никакого значения для NMD не имеют. Таким образом, преждевременные стоп-кодоны располагаются на 50—54 нуклеотида выше, чем последняя граница двух экзонов, а нормальные стоп-кодоны находятся в концевых экзонах[22]. Границы между экзонами отмечают белковые комплексы сращивания экзонов (англ. Exon junction complexes, EJCs). EJC — это многобелковый комплекс, который собирается на транскрипте в ходе сплайсинга на 20—24 нуклеотида выше сайта сплайсинга[23]. Именно благодаря EJC можно отличить преждевременные стоп-кодоны от нормальных. Распознавание преждевременных стоп-кодонов зависит от определения границ между экзонов, поэтому в NMD у млекопитающих задействована сплайсосома[18][24]. Путь NMD не запускается дефектными транскриптами, считанными с генов, которые не содержат интронов, такие как ген гистона H4, Hsp70 и мелакортин-4-рецептора[9].
В норме комплексы EJC находятся после стоп-кодонов. По мере продвижения рибосомы по мРНК она вытесняет комплексы EJC. Когда рибосома доходит до преждевременного стоп-кодона, факторы трансляции eRF1 и eRF3 связываются с невытесненными комплексами EJC, образуя многобелковый мостик[25]. UPF1 взаимодействует с UPF2/UPF3 оставшегося EJC, запуская разрушение мРНК эндогенными нуклеазами[22][25].
У беспозвоночных
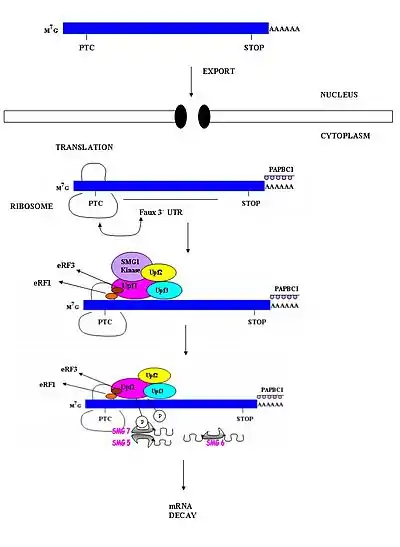
У таких организмов, как S. cerevisiae, D. melanogaster и C. elegans, распознавание стоп-кодонов не связано с границами между экзонами[24], и у них NMD не связан со сплайсингом. По этой причине NMD беспозвоночных не требует участия EJC[4]. Предложено несколько возможных механизмов, позволяющих отличить нормальные стоп-кодоны от преждевременных в клетках беспозвоночных. Согласно одной гипотезе, у них имеется некие последовательности, располагающиеся после преждевременных стоп-кодонов и функционирующие подобно EJC[15]. Вторая модель предполагает, что позиционную информацию, необходимую для различения нормальных и преждевременных стоп-кодонов, могут обеспечивать такие широко распространённые элементы мРНК, как 3'-концевой поли(А)-хвост[26]. Согласно ещё одной модели, 3'-концевые участки, расположенные после нормальных и после преждевременных стоп-кодонов, различаются, например, по связанным с ними белкам. Впрочем, ни одну из этих гипотез пока не удалось подтвердить экспериментально[4].
У растений
У растений имеется два механизма распознавания преждевременных стоп-кодонов: первый связан с расстоянием до EJC, как у позвоночных, а второй опирается на расстояние от стоп-кодона до поли(А)-хвоста. У растений по пути NMD разрушаются мРНК, 3'-нетранслируемая область которых длиннее 300 нуклеотидов, поэтому у растений мРНК с длинными 3'-нетранслируемыми областями встречаются гораздо реже, чем у позвоночных[27][28].
Nonstop-деградация
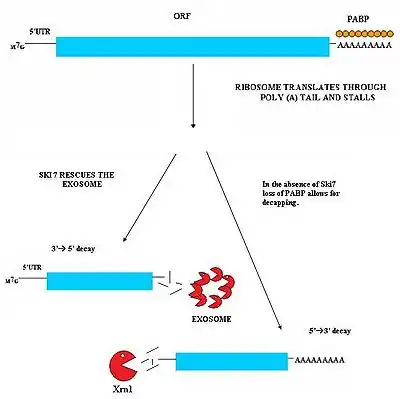
Nonstop-деградация (англ. nonstop mediated decay, NSD) направлена на узнавание и разрушение транскриптов, лишённых стоп-кодонов[30][31]. Такие мРНК могут появиться в результате преждевременного 3'-полиаденилирования, при котором сигналы полиаденилирования находятся в кодирующей области транскрипта[32]. Рибосома, связывающаяся с такими мРНК, осуществляет их трансляцию, пока на она не дойдёт до поли(А)-хвоста, на котором «зависает», и не может диссоциировать от мРНК[33]. Если не избавляться от мРНК без стоп-кодонов, то многие рибосомы будут неспособны транслировать нормальные мРНК, будучи связанными с дефектными транскриптами. Nonstop-деградация высвобождает «зависшие» рибосомы и отправляет мРНК без стоп-кодона на деградацию нуклеазами. Nonstop-деградация протекает по двум основным механизмам, которые, вероятно, действуют совместно[30][31].
Путь Ski7
Белок Ski7, как предполагается, может связываться с пустым А-сайтом рибосомы и тем самым помогает «зависшим» рибосомам освободиться от транскрипта без стоп-кодона. После диссоциации рибосомы Ski7 остаётся связанным с дефектным транскриптом, и именно в таком виде транскрипт разрушается цитозольными экзосомами. Комплекс экзосомы с Ski7 быстро деаденилирует мРНК, и далее экзосома разрушает транскрипт в направлении от 3'-конца к 5'-концу[30][31].
Путь, независимый от Ski7
Второй путь NSD был впервые описан у дрожжей. В отсутствие Ski7 поли(А)-связывающие белки (PABP) диссоциируют от поли(А)-хвоста. Из-за диссоциации белков PABP с транскрипта удаляется защитный 5'-концевой кэп, и транскрипт быстро разрушается эндогенными экзонуклеазами, например, XrnI, направлении от 5'-конца к 3'-концу[31].
No-go-деградация
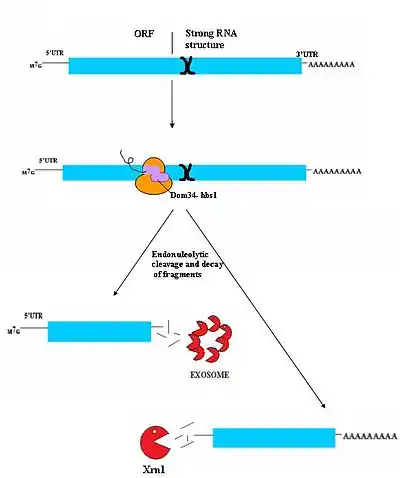
No-go-деградация (англ. No-Go decay, NGD) была описана последней из известных на данный момент механизмов контроля качества мРНК[34], и её механизм ещё не выяснен до конца. Точно неизвестно, какие мРНК являются мишенями NGD, однако предполагается, что это мРНК, на которых в ходе трансляции рибосома «зависла». Это может быть обусловлено вторичной структурой, элементы которой могут физически препятствовать продвижению рибосомы[34]. Комплекс Dom34/Hbs1, вероятно, связывается с мРНК неподалёку от А-сайта «зависшей» рибосомы и помогает ей покинуть транскрипт[35]. В некоторых случаях в транскрипт вносится разрез рядом с тем местом, на котором «зависла» рибосома, однако вносящие этот разрез эндонуклеазы не идентифицированы. Фрагменты транскрипта далее окончательно разрушаются экзосомами в направлении от 3'-конца к 5'-концу или экзонуклеазой Xrn1 в обратном направлении[34]. Не известно, каким именно образом Dom34/Hbs1 способствует диссоциации «зависшей» рибосомы, однако известно, что белок Hbs1 родственен белку Ski7, который выполняет аналогичную роль в nonstop-деградации[7][36].
Эволюция
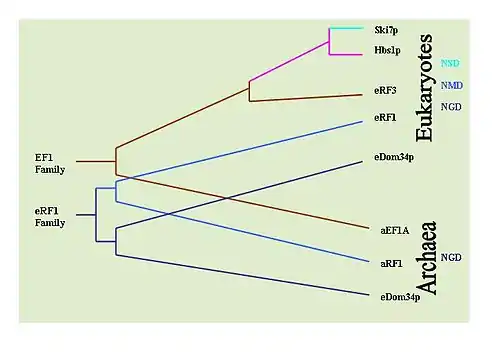
Прослеживая консервативность ключевых белков каждого из механизмов контроля качества мРНК, можно восстановить эволюционную историю этих механизмов. Под ключевыми белками понимаются Dom34/Hbs1 в NGD[34], Ski7 в NSD[30] и eRF в NMD[8]. С помощью BLAST было определено наличие этих белков в разных группах организмов. Оказалось, что Hbs1 (NGD) и eRF3 (NMD) выявляются только у эукариот, а Dom34 (NGD) есть у эукариот и архей. В связи с этим NGD, вероятно, был первым механизмом контроля качества мРНК. Белок Ski7 (NSD) обнаруживается только у дрожжей, поэтому NSD, по всей видимости, появился последним из трёх механизмов. Таким образом, NMD появился вторым из них[37].
Примечания
- Миронова Л. Н., Падкина М. В., Самбук Е. В. РНК: синтез и функции. — СПб.: Эко-вектор, 2017. — С. 272. — 287 с. — ISBN 978-5-906648-29-7.
- Moore M. J. From Birth to Death: The Complex Lives of Eukaryotic mRNAs (англ.) // Science. — 2005. — 2 September (vol. 309, no. 5740). — P. 1514—1518. — ISSN 0036-8075. — doi:10.1126/science.1111443.
- Amrani N., Sachs M. S., Jacobson A. Early nonsense: mRNA decay solves a translational problem. (англ.) // Nature Reviews. Molecular Cell Biology. — 2006. — June (vol. 7, no. 6). — P. 415—425. — doi:10.1038/nrm1942. — PMID 16723977.
- Amrani N., Ganesan R., Kervestin S., Mangus D. A., Ghosh S., Jacobson A. A faux 3'-UTR promotes aberrant termination and triggers nonsense-mediated mRNA decay. (англ.) // Nature. — 2004. — 4 November (vol. 432, no. 7013). — P. 112—118. — doi:10.1038/nature03060. — PMID 15525991.
- Fasken Milo B, Corbett Anita H. Process or perish: quality control in mRNA biogenesis (англ.) // Nature Structural & Molecular Biology. — 2005. — June (vol. 12, no. 6). — P. 482—488. — ISSN 1545-9993. — doi:10.1038/nsmb945.
- Кребс Дж., Голдштейн Э., Килпатрик С. Гены по Льюину. — М.: Лаборатория знаний, 2017. — С. 618. — 919 с. — ISBN 978-5-906828-24-8.
- Chang Y. F., Imam J. S., Wilkinson M. F. The nonsense-mediated decay RNA surveillance pathway. (англ.) // Annual Review Of Biochemistry. — 2007. — Vol. 76. — P. 51—74. — doi:10.1146/annurev.biochem.76.050106.093909. — PMID 17352659.
- Rehwinkel Jan, Raes Jeroen, Izaurralde Elisa. Nonsense-mediated mRNA decay: target genes and functional diversification of effectors (англ.) // Trends in Biochemical Sciences. — 2006. — November (vol. 31, no. 11). — P. 639—646. — ISSN 0968-0004. — doi:10.1016/j.tibs.2006.09.005.
- Maquat Lynne E. Nonsense-mediated mRNA decay: splicing, translation and mRNP dynamics (англ.) // Nature Reviews Molecular Cell Biology. — 2004. — February (vol. 5, no. 2). — P. 89—99. — ISSN 1471-0072. — doi:10.1038/nrm1310.
- Holbrook Jill A, Neu-Yilik Gabriele, Hentze Matthias W, Kulozik Andreas E. Nonsense-mediated decay approaches the clinic (англ.) // Nature Genetics. — 2004. — August (vol. 36, no. 8). — P. 801—808. — ISSN 1061-4036. — doi:10.1038/ng1403.
- Mendell Joshua T, Sharifi Neda A, Meyers Jennifer L, Martinez-Murillo Francisco, Dietz Harry C. Nonsense surveillance regulates expression of diverse classes of mammalian transcripts and mutes genomic noise (англ.) // Nature Genetics. — 2004. — 26 September (vol. 36, no. 10). — P. 1073—1078. — ISSN 1061-4036. — doi:10.1038/ng1429.
- Cali B. M., Kuchma S. L., Latham J., Anderson P. smg-7 is required for mRNA surveillance in Caenorhabditis elegans. (англ.) // Genetics. — 1999. — February (vol. 151, no. 2). — P. 605—616. — PMID 9927455.
- Yamashita Akio, Kashima Isao, Ohno Shigeo. The role of SMG-1 in nonsense-mediated mRNA decay (англ.) // Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. — 2005. — December (vol. 1754, no. 1-2). — P. 305—315. — ISSN 1570-9639. — doi:10.1016/j.bbapap.2005.10.002.
- Kim Yoon Ki, Furic Luc, DesGroseillers Luc, Maquat Lynne E. Mammalian Staufen1 Recruits Upf1 to Specific mRNA 3′UTRs so as to Elicit mRNA Decay (англ.) // Cell. — 2005. — January (vol. 120, no. 2). — P. 195—208. — ISSN 0092-8674. — doi:10.1016/j.cell.2004.11.050.
- Conti Elena, Izaurralde Elisa. Nonsense-mediated mRNA decay: molecular insights and mechanistic variations across species (англ.) // Current Opinion in Cell Biology. — 2005. — June (vol. 17, no. 3). — P. 316—325. — ISSN 0955-0674. — doi:10.1016/j.ceb.2005.04.005.
- Longman D., Plasterk R. H., Johnstone I. L., Cáceres J. F. Mechanistic insights and identification of two novel factors in the C. elegans NMD pathway. (англ.) // Genes & Development. — 2007. — 1 May (vol. 21, no. 9). — P. 1075—1085. — doi:10.1101/gad.417707. — PMID 17437990.
- Gatfield D. Nonsense-mediated mRNA decay in Drosophila:at the intersection of the yeast and mammalian pathways (англ.) // The EMBO Journal. — 2003. — 1 August (vol. 22, no. 15). — P. 3960—3970. — ISSN 1460-2075. — doi:10.1093/emboj/cdg371.
- Nagy E., Maquat L. E. A rule for termination-codon position within intron-containing genes: when nonsense affects RNA abundance. (англ.) // Trends In Biochemical Sciences. — 1998. — June (vol. 23, no. 6). — P. 198—199. — PMID 9644970.
- Inácio Ângela, Silva Ana Luísa, Pinto Joana, Ji Xinjun, Morgado Ana, Almeida Fátima, Faustino Paula, Lavinha João, Liebhaber Stephen A., Romão Luísa. Nonsense Mutations in Close Proximity to the Initiation Codon Fail to Trigger Full Nonsense-mediated mRNA Decay (англ.) // Journal of Biological Chemistry. — 2004. — 25 May (vol. 279, no. 31). — P. 32170—32180. — ISSN 0021-9258. — doi:10.1074/jbc.m405024200.
- Silva A. L., Pereira F. J.C., Morgado A., Kong J., Martins R., Faustino P., Liebhaber S. A., Romao L. The canonical UPF1-dependent nonsense-mediated mRNA decay is inhibited in transcripts carrying a short open reading frame independent of sequence context (англ.) // RNA. — 2006. — 19 October (vol. 12, no. 12). — P. 2160—2170. — ISSN 1355-8382. — doi:10.1261/rna.201406.
- Silva A. L., Ribeiro P., Inacio A., Liebhaber S. A., Romao L. Proximity of the poly(A)-binding protein to a premature termination codon inhibits mammalian nonsense-mediated mRNA decay (англ.) // RNA. — 2008. — 18 January (vol. 14, no. 3). — P. 563—576. — ISSN 1355-8382. — doi:10.1261/rna.815108.
- Zhang Jing, Sun Xiaolei, Qian Yimei, LaDuca Jeffrey P., Maquat Lynne E. At Least One Intron Is Required for the Nonsense-Mediated Decay of Triosephosphate Isomerase mRNA: a Possible Link between Nuclear Splicing and Cytoplasmic Translation (англ.) // Molecular and Cellular Biology. — 1998. — 1 September (vol. 18, no. 9). — P. 5272—5283. — ISSN 0270-7306. — doi:10.1128/mcb.18.9.5272.
- Neu-Yilik G. Splicing and 3' end formation in the definition of nonsense-mediated decay-competent human beta-globin mRNPs (англ.) // The EMBO Journal. — 2001. — 1 February (vol. 20, no. 3). — P. 532—540. — ISSN 1460-2075. — doi:10.1093/emboj/20.3.532.
- Behm-Ansmant I., Gatfield D., Rehwinkel J., Hilgers V., Izaurralde E. A conserved role for cytoplasmic poly(A)-binding protein 1 (PABPC1) in nonsense-mediated mRNA decay. (англ.) // The EMBO Journal. — 2007. — 21 March (vol. 26, no. 6). — P. 1591—1601. — doi:10.1038/sj.emboj.7601588. — PMID 17318186.
- Kashima I., Yamashita A., Izumi N., Kataoka N., Morishita R., Hoshino S., Ohno M., Dreyfuss G., Ohno S. Binding of a novel SMG-1-Upf1-eRF1-eRF3 complex (SURF) to the exon junction complex triggers Upf1 phosphorylation and nonsense-mediated mRNA decay. (англ.) // Genes & Development. — 2006. — 1 February (vol. 20, no. 3). — P. 355—367. — doi:10.1101/gad.1389006. — PMID 16452507.
- Palaniswamy Viswanathan, Moraes Karen C M, Wilusz Carol J, Wilusz Jeffrey. Nucleophosmin is selectively deposited on mRNA during polyadenylation (англ.) // Nature Structural & Molecular Biology. — 2006. — 9 April (vol. 13, no. 5). — P. 429—435. — ISSN 1545-9993. — doi:10.1038/nsmb1080.
- Schwartz A. M., Komarova T. V., Skulachev M. V., Zvereva A. S., Dorokhov IuL., Atabekov J. G. Stability of plant mRNAs depends on the length of the 3'-untranslated region. (англ.) // Biochemistry. Biokhimiia. — 2006. — December (vol. 71, no. 12). — P. 1377—1384. — PMID 17223792.
- Nyikó T., Kerényi F., Szabadkai L., Benkovics A. H., Major P., Sonkoly B., Mérai Z., Barta E., Niemiec E., Kufel J., Silhavy D. Plant nonsense-mediated mRNA decay is controlled by different autoregulatory circuits and can be induced by an EJC-like complex. (англ.) // Nucleic Acids Research. — 2013. — July (vol. 41, no. 13). — P. 6715—6728. — doi:10.1093/nar/gkt366. — PMID 23666629.
- Garneau N. L., Wilusz J., Wilusz C. J. The highways and byways of mRNA decay. (англ.) // Nature Reviews. Molecular Cell Biology. — 2007. — February (vol. 8, no. 2). — P. 113—126. — doi:10.1038/nrm2104. — PMID 17245413.
- van Hoof A., Frischmeyer P. A., Dietz H. C., Parker R. Exosome-mediated recognition and degradation of mRNAs lacking a termination codon. (англ.) // Science (New York, N.Y.). — 2002. — 22 March (vol. 295, no. 5563). — P. 2262—2264. — doi:10.1126/science.1067272. — PMID 11910110.
- Frischmeyer P. A. An mRNA Surveillance Mechanism That Eliminates Transcripts Lacking Termination Codons (англ.) // Science. — 2002. — 22 March (vol. 295, no. 5563). — P. 2258—2261. — ISSN 0036-8075. — doi:10.1126/science.1067338.
- Temperley R. J. Investigation of a pathogenic mtDNA microdeletion reveals a translation-dependent deadenylation decay pathway in human mitochondria (англ.) // Human Molecular Genetics. — 2003. — 15 July (vol. 12, no. 18). — P. 2341—2348. — ISSN 1460-2083. — doi:10.1093/hmg/ddg238.
- Karzai A. W., Roche E. D., Sauer R. T. The SsrA-SmpB system for protein tagging, directed degradation and ribosome rescue. (англ.) // Nature Structural Biology. — 2000. — June (vol. 7, no. 6). — P. 449—455. — doi:10.1038/75843. — PMID 10881189.
- Doma M. K., Parker R. Endonucleolytic cleavage of eukaryotic mRNAs with stalls in translation elongation. (англ.) // Nature. — 2006. — 23 March (vol. 440, no. 7083). — P. 561—564. — doi:10.1038/nature04530. — PMID 16554824.
- Kobayashi K., Kikuno I., Kuroha K., Saito K., Ito K., Ishitani R., Inada T., Nureki O. Structural basis for mRNA surveillance by archaeal Pelota and GTP-bound EF1α complex. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2010. — 12 October (vol. 107, no. 41). — P. 17575—17579. — doi:10.1073/pnas.1009598107. — PMID 20876129.
- Graille M., Chaillet M., van Tilbeurgh H. Structure of yeast Dom34: a protein related to translation termination factor Erf1 and involved in No-Go decay. (англ.) // The Journal Of Biological Chemistry. — 2008. — 14 March (vol. 283, no. 11). — P. 7145—7154. — doi:10.1074/jbc.M708224200. — PMID 18180287.
- Atkinson G. C., Baldauf S. L., Hauryliuk V. Evolution of nonstop, no-go and nonsense-mediated mRNA decay and their termination factor-derived components. (англ.) // BMC Evolutionary Biology. — 2008. — 23 October (vol. 8). — P. 290—290. — doi:10.1186/1471-2148-8-290. — PMID 18947425.