Гемагглютинин вируса гриппа
Гемагглютинин вируса гриппа (HA) — гомотримерный гликопротеин, обнаруживаемый на поверхности вируса гриппа и являющийся ключевым для его механизма заражения.
| Гемагглютинин | |
|---|---|
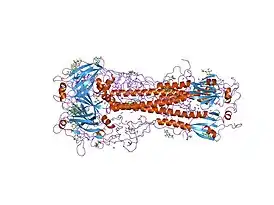 | |
| Идентификаторы | |
| Символ | Гемагглютинин |
| Pfam | PF00509 |
| InterPro | IPR001364 |
| SCOP | 1hgd |
| SUPERFAMILY | 1hgd |
| OPM superfamily | 109 |
| OPM protein | 6hjq |
| Доступные структуры белков | |
| Pfam | структуры |
| PDB | RCSB PDB; PDBe; PDBj |
| PDBsum | 3D-модель |
| Стебель гемагглютинина вируса гриппа подтипа C | |
|---|---|
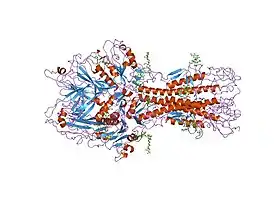 Структура гликопротеина слияния гемагглютинина и эстеразы вируса гриппа С, полученная путём рентгеновской кристаллографии | |
| Идентификаторы | |
| Символ | Hema_stalk |
| Pfam | PF08720 |
| InterPro | IPR014831 |
| SCOP | 1flc |
| SUPERFAMILY | 1flc |
| OPM superfamily | 277 |
| OPM protein | 2jrd |
| Доступные структуры белков | |
| Pfam | структуры |
| PDB | RCSB PDB; PDBe; PDBj |
| PDBsum | 3D-модель |
Гемагглютинин — белок слияния класса I, помимо функции присоединения к мембране клетки, в дальнейшем также непосредственно осуществляющий слияние мембран. Таким образом, гемагглютинин, с одной стороны, отвечает за присоединение вируса гриппа к молекуле сиаловой кислоты на мембране клетки-мишени — в частности, клеток верхних дыхательных путей или эритроцитов[1], с последующим эндоцитозом вируса[2]. Далее, оказываясь в среде с низким pH (5.0-5.5), гемагглютинин вызывает слияние оболочки вируса с мембранной эндосомой[3].
Название «гемагглютинин» происходит от способности этого белка приводить к слипанию (агглютинации) красных кровяных клеток (эритроцитов) in vitro[4].
Подтипы
Известно не менее 18 различных разновидностей гемагглютинина вируса гриппа типа A. Эти подтипы маркируются обозначениями от H1 до H18. Подтип H16 был идентифицирован в 2004 году в вирусах гриппа, обнаруженных у черноголовых чаек в Швеции и Норвегии; подтип H17 — в 2012 году у летучих мышей[5][6]. Подтип H18 был обнаружен у летучей мыши из Перу в 2013 году[7]. Первые три подтипа, H1, H2, and H3, встречаются в человеческих вирусах гриппа. По степени филогенетической схожести гемагглютинины гриппа делятся на две группы: в первую входят подтипы H1, H2, H5, H6, H8, H9, H11, H12, H13, H16, H17 и H18, а во вторую — все остальные[8]. Серотип вируса гриппа типа A определяется по подтипам находящихся на его поверхности гемагглютинина и нейраминидазы[9]. Нейраминидаза (NA) имеет 11 известных подтипов; соответственно, серотип вируса обозначается как H1N1, H5N2 и т. д.
Высокопатогенный птичий грипп серотипа H5N1 оказался способным заражать людей, с невысокой частотой. Согласно сообщениям, у пациентов было найдено единственное изменение в аминокислотной последовательности гемагглютинина H5, которое «может значительно изменить специфичность к рецепторам у птичьих вирусов H5N1, тем самым давая им возможность присоединяться к рецепторам, оптимальным для вирусов гриппа человека»[10][11]. Эта находка, возможно, объясняет, как птичий вирус H5N1, который обычно не инфицирует людей, может мутировать и эффективно заражать клетки человека. Гемагглютинин вируса H5N1 был ассоциирован с высокой патогенностью этого птичьего штамма — вероятно, благодаря лёгкости его преобразования в активную форму посредством протеолиза[12][13].
Структура
Гемагглютинин — гомотримерный мембранный гликопротеин. Он имеет форму цилиндра длиной около 13,5 нанометра[14][15]. Тример гемагглютинина состоит из трёх идентичных мономеров, каждый из которых, в свою очередь, представляет собой одиночную полипептидную цепь HA0 с субъединицами HA1 и HA2, связанными двумя дисульфидными мостиками[15][16]. Каждая из областей HA2 представляет собой структуру из альфа-спиралей, которая располагается поверх области HA1, представляющей собой небольшой округлый домен из нескольких α/β структур[17].
Тример гемагглютинина синтезируется в виде неактивного белка-предшественника HA0 во избежание нежелательного преждевременного слияния, и, чтобы стать активным, должен быть расщеплен при помощи протеаз клетки-хозяина. При нейтральных значениях pH, 23 остатка поблизости от N-конца субъединицы HA2 (также известные как пептид слияния, ответственный за соединение мембран вируса и клетки-хозяина) спрятаны в гидрофобном «кармане» между областями взаимодействия тримера HA2[18]. C-конец субъединицы HA2, также известный как трансмембранный домен, проходит сквозь капсид вируса и фиксирует на нём белки[19].
- HA1
- Субъединица HA1 большей частью состоит из антипараллельных бета-листов[14].
- HA2
- Субъединица HA2 содержит три длинных альфа-спирали, по одной от каждого мономера. Каждая из этих спиралей присоединена при помощи длинного кольцевого региона, называемого «B-кольцом» (остатки 59-76)[20].
Функции
Гемагглютинин играет две ключевые роли в проникновении вируса в клетку. Во-первых, он распознаёт клетки-мишени позвоночных, присоединяясь к рецепторам этих клеток, содержащим сиаловую кислоту. Во-вторых, после захвата вируса в эндосому клетки, он обеспечивает проникновение в клетку вирусного генома, обеспечивая слияние мембраны эндосомы с капсидом вируса[21].
В частности, его субъединица HA1 соединяется с моносахаридом — сиаловой кислотой, — присутствующим на поверхности его клеток-мишеней, тем самым прикрепляя вирион к поверхности клетки-хозяина. Было показано, что варианты HA17 и HA18 в качестве рецептора-цели используют не сиаловую кислоту, а молекулы главного комплекса гистосовместимости второго типа[22]. Далее мембрана клетки-хозяина поглощает вирус путём эндоцитоза, и отслаивается, образуя новый мембраносвязанный отсек внутри клетки, называемый эндосомой. Затем клетка пытается уничтожить содержимое эндосомы, подкисляя её содержимое и превращая её в лизосому. Когда pH внутри эндосомы достигает уровня между 5.0 и 6.0, гемагглютинин претерпевает ряд конформационных изменений. Прежде всего, пептид слияния высвобождается из гидрофобного «кармана», а субъединица HА1 отсоединяется от субъединицы HА2. Затем субъединица HА2 претерпевает ряд значительных конформационных изменений, в результате которых мембраны вируса и эндосомы сближаются.
Пептид слияния, высвобожденный ранее пониженным уровнем рН, действует как молекулярный «абордажный крюк», внедряясь в мембрану эндосомы и закрепляясь в ней. Затем субъединица HA2 вновь изменяет конформацию (которая более стабильна при более низком рН), втягивая «абордажный крюк», тем самым приближая мембрану эндосомы вплотную к мембране самого вируса, в результате чего обе мембраны сливаются. Как результат, содержимое вирусной частицы в виде вирусной РНК высвобождается в цитоплазме клетки-хозяина, а затем транспортируется в ядро клетки-хозяина для репликации[23].
Гемагглютинин вируса как цель для лекарственных препаратов
Поскольку гемагглютинин играет ключевую роль в процессе проникновения вируса гриппа A в клетку, он является основной целью для нейтрализующих антител. Было показано, что такие антитела действуют по одному из нижеследующих механизмов, соответствующим каждой из функций гемагглютинина:
Антитела к головному участку
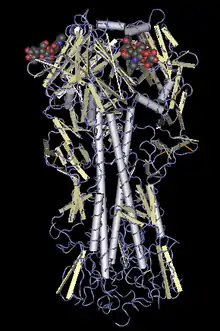
Некоторые антитела к гемагглютинину подавляют его прикрепление к клетке. Такие антитела прикрепляются вблизи «головки» гемагглютинина (выделена синим на рисунке выше) и физически не допускают его взаимодействия с рецепторами сиаловой кислоты на клетках-мишенях[24].
Стволовые антитела
Эта группа антител действует, препятствуя слиянию мембран вируса и эндосомы (только in vitro; эффективность таких антител in vivo считается результатом действия клеток иммунной системы, а также системы комлемента)[25].
Область ствола или «стебля» субъединицы HA2 во многом высоко консервативна среди различных штаммов вирусов гриппа. Такая консервативность делает её привлекательной целью для нейтрализующих антител, которые смогут воздействовать на все подтипы вирусов гриппа, а также для разработки универсальной вакцины, которая позволит вырабатывать такие антитела естественным путём[26]. Структурные изменения субъединицы HA2 от префузионной к постфузионной конформации приводят к слиянию вирусной мембраны с мембраной хозяина. Антитела, прикрепляющиеся к этой единице, могут помешать процессу слияния мембран, и тем самым заблокировать важную стадию жизненного цикла сразу многих серотипов вируса гриппа. По крайней мере одно рассмотренное антитело прикрепляется ближе к «голове» гемагглютинина, и считается, что оно действует, путем сшивания головок мономеров вместе, открытие которых считается первым шагом в процессе слияния мембран[27].
Примерами таких антител являются человеческие антитела F10[28], FI6[29], CR6261. Они распознают сайты в области «стебля» гемагглютинина (область, выделенная на рисунке оранжевым), находящиеся на значительном удалении от сайта связывания с рецептором[30][31].
В 2015 году исследователи разработали иммуноген, имитирующий ствол гемагглютинина, а конкретнее — его область, к которой присоединяется антитело CR9114. Эксперименты на модельных грызунах и приматах, которым вводился этот иммуноген, привели к появлению у них антител, способных соединятся с гемагглютининами многих подтипов гриппа, включая H5N1[32]. В случае присутствия «головки» гемагглютинина иммунная система в общем случае не создаёт широко нейтрализующие антитела; вместо этого, она изготавливает антитела, которые «распознают» всего несколько подтипов вируса. Поскольку «головка» удерживает все три субъединицы гемагглютинина сложенными вместе, «только-стволовый» гемагглютинин нуждается в собственном способе удержания субъединиц. Одна команда учёных разработала самосборные наночастицы ствола гемагглютинина, используя белок, называемый ферритином, для удержания субъединиц гемагглютинина вместе. Другая команда использовала замену и добавление аминокислот для стабилизации «мини-гемагглютинина», у которого отсутствует собственная головка.
В экспериментах 2016 года был обнаружен ряд антител, произведённых иммунной системой человека, «настроенных» на присоединение к стволу. От ряда добровольцев были получены три класса похожих антител, что подтолкнуло исследователей к выводу, что создание универсальной вакцины, приводящей к надёжному созданию универсальных антител, всё-таки возможно[33].
См. также
Примечания
- Russell RJ, Kerry PS, Stevens DJ, Steinhauer DA, Martin SR, Gamblin SJ, Skehel JJ (November 2008). “Structure of influenza Hemagglutinin in complex with an inhibitor of membrane fusion”. Proceedings of the National Academy of Sciences of the United States of America. 105 (46): 17736—41. DOI:10.1073/pnas.0807142105. PMC 2584702. PMID 19004788.
- Edinger, Thomas O.; Pohl, Marie O.; Stertz, Silke (February 2014). “Entry of influenza A virus: host factors and antiviral targets” (PDF). The Journal of General Virology. 95 (Pt 2): 263—277. DOI:10.1099/vir.0.059477-0. ISSN 1465-2099. PMID 24225499.
- Horvath, Peter; Helenius, Ari; Yamauchi, Yohei; Banerjee, Indranil (2013-07-12). “High-Content Analysis of Sequential Events during the Early Phase of Influenza A Virus Infection”. PLOS ONE. 8 (7): e68450. DOI:10.1371/journal.pone.0068450. ISSN 1932-6203. PMC 3709902. PMID 23874633.
- Lehninger's Principles of Biochemistry. — 4th. — New York : WH Freeman, 2005.
- Fouchier RA, Munster V, Wallensten A, Bestebroer TM, Herfst S, Smith D, Rimmelzwaan GF, Olsen B, Osterhaus AD (March 2005). “Characterization of a novel influenza A virus hemagglutinin subtype (H16) obtained from black-headed gulls”. Journal of Virology. 79 (5): 2814—22. DOI:10.1128/JVI.79.5.2814-2822.2005. PMC 548452. PMID 15709000.
- Unique new flu virus found in bats.
- Tong S, Zhu X, Li Y, Shi M, Zhang J, Bourgeois M, et al. (October 2013). “New world bats harbor diverse influenza A viruses”. PLOS Pathogens. 9 (10): e1003657. DOI:10.1371/journal.ppat.1003657. PMC 3794996. PMID 24130481.
- Sutton, Troy C.; Chakraborty, Saborni; Mallajosyula, Vamsee V. A.; Lamirande, Elaine W.; Ganti, Ketaki; Bock, Kevin W.; Moore, Ian N.; Varadarajan, Raghavan; Subbarao, Kanta (15 December 2017). “Protective efficacy of influenza group 2 hemagglutinin stem-fragment immunogen vaccines”. NPJ Vaccines. 2 (1): 35. DOI:10.1038/s41541-017-0036-2. PMC 5732283. PMID 29263889.
- Influenza Type A Viruses. Avian Influenza (Flu). CDC (19 апреля 2017). Дата обращения: 27 августа 2018.
- Suzuki Y (March 2005). “Sialobiology of influenza: molecular mechanism of host range variation of influenza viruses”. Biological & Pharmaceutical Bulletin. 28 (3): 399—408. DOI:10.1248/bpb.28.399. PMID 15744059.
- Gambaryan A, Tuzikov A, Pazynina G, Bovin N, Balish A, Klimov A (January 2006). “Evolution of the receptor binding phenotype of influenza A (H5) viruses”. Virology. 344 (2): 432—8. DOI:10.1016/j.virol.2005.08.035. PMID 16226289.
- Hatta M, Gao P, Halfmann P, Kawaoka Y (September 2001). “Molecular basis for high virulence of Hong Kong H5N1 influenza A viruses”. Science. 293 (5536): 1840—2. DOI:10.1126/science.1062882. PMID 11546875.
- Senne DA, Panigrahy B, Kawaoka Y, Pearson JE, Süss J, Lipkind M, Kida H, Webster RG (1996). “Survey of the hemagglutinin (HA) cleavage site sequence of H5 and H7 avian influenza viruses: amino acid sequence at the HA cleavage site as a marker of pathogenicity potential”. Avian Diseases. 40 (2): 425—37. DOI:10.2307/1592241. JSTOR 1592241. PMID 8790895.
- Wilson IA, Skehel JJ, Wiley DC (January 1981). “Structure of the haemagglutinin membrane glycoprotein of influenza virus at 3 A resolution”. Nature. 289 (5796): 366—73. DOI:10.1038/289366a0. PMID 7464906.
- Boonstra S, Blijleven JS, Roos WH, Onck PR, van der Giessen E, van Oijen AM (May 2018). “Hemagglutinin-Mediated Membrane Fusion: A Biophysical Perspective”. Annual Review of Biophysics. 47 (1): 153—173. DOI:10.1146/annurev-biophys-070317-033018. PMID 29494252.
- Di Lella S, Herrmann A, Mair CM (June 2016). “Modulation of the pH Stability of Influenza Virus Hemagglutinin: A Host Cell Adaptation Strategy”. Biophysical Journal. 110 (11): 2293—2301. DOI:10.1016/j.bpj.2016.04.035. PMC 4906160. PMID 27276248.
- Smrt, Sean T. & Lorieau, Justin L. (2016), Membrane Fusion and Infection of the Influenza Hemagglutinin, Advances in Experimental Medicine and Biology (Springer Singapore) . — Т. 966: 37–54, ISBN 9789811069215, PMID 27966108, DOI 10.1007/5584_2016_174
- Wiley DC, Skehel JJ (June 1987). “The structure and function of the hemagglutinin membrane glycoprotein of influenza virus”. Annual Review of Biochemistry. 56 (1): 365—94. DOI:10.1146/annurev.bi.56.070187.002053. PMID 3304138.
- H., Strauss, James. Viruses and human disease. — 2nd. — Amsterdam : Elsevier / Academic Press, 2008. — ISBN 9780080553160.
- Stevens J, Corper AL, Basler CF, Taubenberger JK, Palese P, Wilson IA (March 2004). “Structure of the uncleaved human H1 hemagglutinin from the extinct 1918 influenza virus”. Science. 303 (5665): 1866—70. DOI:10.1126/science.1093373. PMID 14764887.
- Attachment and entry of influenza virus into host cells. Pivotal roles of hemagglutinin // Structural Biology of Viruses. — Oxford University Press, 1997. — P. 80–104.
- Karakus, Umut; Thamamongood, Thiprampai; Ciminski, Kevin; Ran, Wei; Günther, Sira C.; Pohl, Marie O.; Eletto, Davide; Jeney, Csaba; Hoffmann, Donata (March 2019). “MHC class II proteins mediate cross-species entry of bat influenza viruses”. Nature. 567 (7746): 109—112. DOI:10.1038/s41586-019-0955-3. ISSN 1476-4687. PMID 30787439.
- Mair CM, Ludwig K, Herrmann A, Sieben C (April 2014). “Receptor binding and pH stability - how influenza A virus hemagglutinin affects host-specific virus infection”. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (4): 1153—68. DOI:10.1016/j.bbamem.2013.10.004. PMID 24161712.
- Goh BC, Rynkiewicz MJ, Cafarella TR, White MR, Hartshorn KL, Allen K, Crouch EC, Calin O, Seeberger PH, Schulten K, Seaton BA (November 2013). “Molecular mechanisms of inhibition of influenza by surfactant protein D revealed by large-scale molecular dynamics simulation”. Biochemistry. 52 (47): 8527—38. DOI:10.1021/bi4010683. PMC 3927399. PMID 24224757.
- DiLillo DJ, Tan GS, Palese P, Ravetch JV (February 2014). “Broadly neutralizing hemagglutinin stalk-specific antibodies require FcγR interactions for protection against influenza virus in vivo”. Nature Medicine. 20 (2): 143—51. DOI:10.1038/nm.3443. PMC 3966466. PMID 24412922.
- Sautto GA, Kirchenbaum GA, Ross TM (January 2018). “Towards a universal influenza vaccine: different approaches for one goal”. Virology Journal. 15 (1): 17. DOI:10.1186/s12985-017-0918-y. PMC 5785881. PMID 29370862.
- Barbey-Martin C, Gigant B, Bizebard T, Calder LJ, Wharton SA, Skehel JJ, Knossow M (March 2002). “An antibody that prevents the hemagglutinin low pH fusogenic transition”. Virology. 294 (1): 70—4. DOI:10.1006/viro.2001.1320. PMID 11886266.
- Sui J, Hwang WC, Perez S, Wei G, Aird D, Chen LM, Santelli E, Stec B, Cadwell G, Ali M, Wan H, Murakami A, Yammanuru A, Han T, Cox NJ, Bankston LA, Donis RO, Liddington RC, Marasco WA (March 2009). “Structural and functional bases for broad-spectrum neutralization of avian and human influenza A viruses”. Nature Structural & Molecular Biology. 16 (3): 265—73. DOI:10.1038/nsmb.1566. PMC 2692245. PMID 19234466.
- Corti D, Voss J, Gamblin SJ, Codoni G, Macagno A, Jarrossay D, Vachieri SG, Pinna D, Minola A, Vanzetta F, Silacci C, Fernandez-Rodriguez BM, Agatic G, Bianchi S, Giacchetto-Sasselli I, Calder L, Sallusto F, Collins P, Haire LF, Temperton N, Langedijk JP, Skehel JJ, Lanzavecchia A (August 2011). “A neutralizing antibody selected from plasma cells that binds to group 1 and group 2 influenza A hemagglutinins”. Science. 333 (6044): 850—6. DOI:10.1126/science.1205669. PMID 21798894.
- Throsby M, van den Brink E, Jongeneelen M, Poon LL, Alard P, Cornelissen L, Bakker A, Cox F, van Deventer E, Guan Y, Cinatl J, ter Meulen J, Lasters I, Carsetti R, Peiris M, de Kruif J, Goudsmit J (2008). “Heterosubtypic neutralizing monoclonal antibodies cross-protective against H5N1 and H1N1 recovered from human IgM+ memory B cells”. PLOS ONE. 3 (12): e3942. DOI:10.1371/journal.pone.0003942. PMC 2596486. PMID 19079604.
- Ekiert DC, Bhabha G, Elsliger MA, Friesen RH, Jongeneelen M, Throsby M, Goudsmit J, Wilson IA (April 2009). “Antibody recognition of a highly conserved influenza virus epitope”. Science. 324 (5924): 246—51. DOI:10.1126/science.1171491. PMC 2758658. PMID 19251591.
- MICU, ALEXANDRU Universal flu vaccine: research moves closer. ZME Science (25 августа 2015). Дата обращения: 10 июня 2016.
- Joyce, MG; Wheatley, AK; Thomas, PV; Chuang, GY; Soto, C; Bailer, RT; Druz, A; Georgiev, IS; Gillespie, RA; Kanekiyo, M; Kong, WP; Leung, K; Narpala, SN; Prabhakaran, MS; Yang, ES; Zhang, B; Zhang, Y; Asokan, M; Boyington, JC; Bylund, T; Darko, S; Lees, CR; Ransier, A; Shen, CH; Wang, L; Whittle, JR; Wu, X; Yassine, HM; Santos, C; Matsuoka, Y; Tsybovsky, Y; Baxa, U; NISC Comparative Sequencing, Program.; Mullikin, JC; Subbarao, K; Douek, DC; Graham, BS; Koup, RA; Ledgerwood, JE; Roederer, M; Shapiro, L; Kwong, PD; Mascola, JR; McDermott, AB (2016-07-28). “Vaccine-Induced Antibodies that Neutralize Group 1 and Group 2 Influenza A Viruses”. Cell. 166 (3): 609—623. DOI:10.1016/j.cell.2016.06.043. PMC 4978566. PMID 27453470.
- Zeng LY, Yang J, Liu S (January 2017). “Investigational hemagglutinin-targeted influenza virus inhibitors”. Expert Opinion on Investigational Drugs. 26 (1): 63—73. DOI:10.1080/13543784.2017.1269170. PMID 27918208.