R-петля
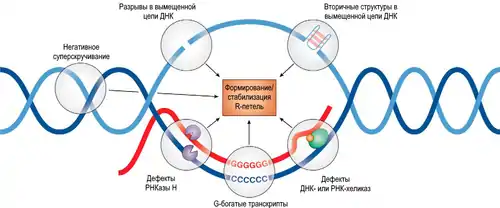
R-петля — особая структурная конформация нуклеиновых кислот, образующаяся при гибридизации РНК с комплементарной нитью двунитевой ДНК, образование дуплекса РНК:ДНК при этом приводит к вытеснению некомплементарной нити ДНК с образованием петли[1]. R-петли часто формируются во время транскрипции, особенно в промоторных областях, когда только что синтезированный транскрипт гибридизуется с матричной цепью ДНК[2]. По некоторым оценкам R-петли могут занимать до 5 % генома у млекопитающих. R-петли играют роль в стабильности генома и хорошо известных функциях, связанных с регуляцией генов, репликацией ДНК, формированием паттерна хроматина, рекомбинацией генов иммуноглобулинов и репарацией двухцепочечных разрывов ДНК[3][4].
История
R-петли были впервые идентифицированы in vitro при помощи электронной микроскопии в 1976 году[5]. В эксперименте in vitro было продемонстрировало образование стабильной гибридных структур РНК с двунитевой ДНК, для разрушения этих структур требовалась деградация РНК при помощи РНКаз. Однако эксперименты по гибридизации in vitro требовали использования 70 % формамида, и поэтому не было доказано, что наблюдаемые структуры встречаются в природе. Тогда было высказано предположение, что при помощи R-петель можно картировать и выявлять последовательности ДНК. Два десятилетия спустя аналогичные гибридные структуры РНК:ДНК были идентифицированы in vivo. В этом исследовании мутантный анализ в Escherichia coli идентифицировал субъединицу ДНК-гиразы В и показал, что сверхэкспрессия эндорибонуклеазы Н (РНКазы Н) и топоизомеразы I в качестве ключевых ферментов сбалансированного образования R-петель уменьшает дефекты роста, вызванные накоплением R-петель. С момента этого открытия биологическая роль R-петель в отношении транскрипции / регуляции генов, рекомбинации с переключением классов (CSR)/диверсификации классов антител и стабильности репарации ДНК/генома рассматривалась в большом количестве комплексных исследований[6].
Последние исследования
В ходе исследований были проанализированы крупномасштабные наборы данных о белках, ассоциированных с гибридной петлей, и данные об экспрессии генов из базы данных генома рака.[3] Исследователи предполагают, что РНК:гибридные белки ДНК являются релевантными маркерами и мишенями для терапии рака. Аналогичным образом, различные белки, связанные с регуляцией эпитранскриптома, также способствуют развитию раковых явлений, а METTL3 (m6 A writer) и YTHDF2 (m6 A reader), которые, как было показано, играют роль в биологии R — петли, были связаны с развитием рака в нескольких исследованиях. Роль m6A в биологии R-петли и возможной стабильности генома является очень недавним открытием, и модификации M6a на мРНК имеют несколько функций, включая стабильность мРНК, транспорт и сплайсинг. Таким образом, неизвестно, относится ли биологическая роль модифицированных мРНК M6a к отдельным мРНК или к их гибридизации с ДНК в структурах R-петли. В дополнение к раку, структуры R-петли также были вовлечены в нарушения повторного расширения и многочисленные неврологические заболевания. В мозге происходит динамическое метилирование м6А в мРНК, и механизм, контролирующий это, необходим для точной нейрорегуляции. Опять же, эпитранскриптомика показала, что белки, влияющие на формирование и накопление R-петель, коррелируют с дефектом развития мозга, самообновлением нейрональных стволовых клеток-предшественников и развитием нейронов у мышей. Интересно, что истощение YTHDF2 у мышей приводит к накоплению структур R-петли и очагов yH2AX в коре плода. Формирование, функция и разрешение R-петель в геноме млекопитающих широко изучались в течение последних десятилетий, но некоторые интересные свойства, связанные со стабильностью генома и болезнями, ещё предстоит понять[4][3][7][8][9].
Примечания
- Забудская К. R-петли: новый штрих нестабильности генома. https://medach.pro/. Medical Channel (25.10.2019). Дата обращения: 19 июня 2021.
- Hegazy Y. A., Fernando C. M., Tran E. J. The balancing act of R-loop biology: The good, the bad, and the ugly (англ.) // Journal of Biological Chemistry. — 2020. — Vol. 295, no. 4. — P. 905-913. — doi:10.1016/S0021-9258(17)49903-0.
- Boros-Ol ah, et al., Drugging the R-loop interactome: RNA-DNA hybrid binding proteins as targets for cancer therapy, DNA Repair 84 (2019) 102642
- J. Paris, et al., Targeting the RNA m(6)A reader YTHDF2 selectively compromises Cancer stem cells in acute myeloid leukemia, Cell Stem Cell 25 (1) (2019) 137—148.e6
- Groh M., Gromak N. Out of balance: R-loops in human disease (англ.) // PLoS Genet. — 2014. — Vol. 10, no. 9. — P. e1004630. — doi:10.1371/journal.pgen.1004630.
- Modifications and interactions at the R-loop (англ.) // DNA Repair. — 2020-12-01. — Vol. 96. — P. 102958. — ISSN 1568-7864. — doi:10.1016/j.dnarep.2020.102958.
- Haiyun Xie, Jiangfeng Li, Yufan Ying, Huaqing Yan, Ke Jin. METTL3/YTHDF2 m6A axis promotes tumorigenesis by degrading SETD7 and KLF4 mRNAs in bladder cancer // Journal of Cellular and Molecular Medicine. — 2020-03-03. — Т. 24, вып. 7. — С. 4092–4104. — ISSN 1582-4934 1582-1838, 1582-4934. — doi:10.1111/jcmm.15063.
- Shuibin Lin, Junho Choe, Peng Du, Robinson Triboulet, Richard I. Gregory. The m 6 A Methyltransferase METTL3 Promotes Translation in Human Cancer Cells // Molecular Cell. — 2016-05. — Т. 62, вып. 3. — С. 335–345. — ISSN 1097-2765. — doi:10.1016/j.molcel.2016.03.021.
- Patricia Richard, James L. Manley. R Loops and Links to Human Disease // Journal of Molecular Biology. — 2017-10. — Т. 429, вып. 21. — С. 3168–3180. — ISSN 0022-2836. — doi:10.1016/j.jmb.2016.08.031.