Bioconductor
Bioconductor — это масштабный FLOSS-проект, предоставляющий множество отдельных пакетов для биоинформатических исследований. Включает набор инструментов для анализа и понимания геномных данных, аннотированные имена чипов, аннотации биологических последовательностей и т. д., а также сами экспериментальные данные.
| Bioconductor | |
|---|---|
 | |
 | |
| Тип | Биоинформатика |
| Операционная система | Linux, Mac OS X, Windows |
| Языки интерфейса | R |
| Последняя версия | 3.6 |
| Лицензия | Artistic License 2.0 |
| Сайт | bioconductor.org |
Использует язык программирования R, благодаря чему поддерживает кросплатформенность (Linux, большинство UNIX-подобных систем, Mac OS X, Windows). Разрабатывается и распространяется свободно и открыто. Выпуск релизов происходит дважды в год. Обладает активным свободным сообществом разработчиков и учёных, что свойственно хорошим открытым проектам. Описания проекта и изменений в нём выпускаются в виде ежегодного отчёта[1].
На данный момент содержит более 2000 пакетов.
Цели проекта
Основными целями Bioconductor’а являются:
- Обеспечение открытого доступа к широкому кругу мощных статистических и графических методов анализа геномных данных.
- Облегчение включения биологических метаданных в анализ геномных данных, например, литературных данных с PubMed, аннотированных данных с Entrez.
- Обеспечение общей программной платформы, которая позволяет быстро разрабатывать и развёртывать расширяемые, масштабируемые и совместимые программы.
- Дальнейшее научное понимание при создании высококачественной документации для воспроизводимых исследований.
- Обучение исследователей вычислительным и статистическим методам анализа геномных данных.
История проекта
Начало проекта было положено в 2001-м году в Dana Farber Cancer Institute.[2]
Пакеты
Три основных группы пакетов — это собственно статистические и графические пакеты (Software), аннотационные пакеты (AnnotationData) и экспериментальные данные (ExperimentData).
В скобках указано количество пакетов в группе.
- aCGH (12)
- CellBasedAssays (38)
- ChIPchip (7)
- CopyNumberVariation (43)
- CpGIsland (6)
- DNAMethylation (38)
- ExonArray (6)
- GeneExpression (131)
- GeneticVariability (23)
- SNP (40)
- Transcription (47)
- AlternativeSplicing (7)
- Coverage (13)
- DifferentialExpression (164)
- DifferentialMethylation (8)
- DifferentialPeakCalling (1)
- DifferentialSplicing (6)
- FunctionalPrediction (1)
- GeneRegulation (21)
- GeneSetEnrichment (41)
- GeneTarget (1)
- GenomeAnnotation (10)
- GenomicVariation (6)
- LinkageDisequilibrium (1)
- MotifAnnotation (3)
- MotifDiscovery (4)
- NetworkEnrichment (13)
- NetworkInference (17)
- SequenceMatching (17)
- SomaticMutation (4)
- VariantDetection (1)
- DataImport (82)
- DataRepresentation (34)
- GUI (19)
- ThirdPartyClient (9)
- BiomedicalInformatics (2)
- CellBiology (20)
- Cheminformatics (3)
- FunctionalGenomics (1)
- Genetics (96)
- Metabolomics (12)
- Metagenomics (5)
- Proteomics (65)
- SystemsBiology (10)
- Bayesian (15)
- Classification (65)
- Clustering (89)
- DecisionTree (7)
- DimensionReduction (2)
- FeatureExtraction (2)
- GraphAndNetwork (69)
- HiddenMarkovModel (4)
- NeuralNetwork (1)
- PrincipalComponent (2)
- Regression (7)
- StructuralEquationModels (1)
- SupportVectorMachine (1)
- Survival (4)
- TimeCourse (29)
- FlowCytometry (36)
- ChipOnChip (1)
- MethylationArray (7)
- mRNAMicroarray (3)
- MultiChannel (3)
- OneChannel (68)
- ProprietaryPlatforms (2)
- TwoChannel (53)
- MicrotitrePlateAssay (13)
- qPCR (9)
- SAGE (9)
- ChIPSeq (40)
- DNASeq (6)
- ExomeSeq (3)
- MethylSeq (10)
- Microbiome (3)
- miRNA (4)
- RIPSeq (1)
- RNASeq (76)
- TargetedResequencing (2)
- WholeGenome (3)
- Alignment (8)
- Annotation (67)
- BatchEffect (5)
- ExperimentalDesign (2)
- MultipleComparison (76)
- Normalization (15)
- GO (33)
- Preprocessing (128)
- QualityControl (95)
- ReportWriting (25)
- Network (44)
- AffymetrixChip (336)
- AgilentChip (15)
- IlluminaChip (19)
- INDACChip (1)
- QiagenChip (1)
- RNG_MRCChip (2)
- adme16cod (1)
- ag (3)
- ath1121501 (3)
- celegans (3)
- drosgenome1 (3)
- drosophila2 (3)
- h10kcod (1)
- h20kcod (1)
- hcg110 (3)
- hgfocus (3)
- hgu133a (4)
- hgu133a2 (4)
- hgu133b (3)
- hgu133plus2 (4)
- hgu95a (3)
- hgu95av2 (4)
- hgu95b (3)
- hgu95c (3)
- hgu95d (3)
- hgu95e (3)
- hguatlas13k (1)
- hgug4100a (1)
- hgug4101a (1)
- hgug4110b (1)
- hgug4111a (1)
- hgug4112a (1)
- hguqiagenv3 (1)
- hi16cod (1)
- hs25kresogen (1)
- hu35ksuba (3)
- hu35ksubb (3)
- hu35ksubc (3)
- hu35ksubd (3)
- hu6800 (3)
- HuO22 (1)
- hwgcod (1)
- illuminaHumanv1 (1)
- illuminaHumanv2 (1)
- illuminaMousev1 (1)
- illuminaMousev1p1 (1)
- illuminaRatv1 (1)
- indac (1)
- m10kcod (1)
- m20kcod (1)
- mgu74a (3)
- mgu74av2 (3)
- mgu74b (3)
- mgu74bv2 (3)
- mgu74c (3)
- mgu74cv2 (3)
- mguatlas5k (1)
- mgug4121a (1)
- mgug4122a (1)
- mi16cod (1)
- mm24kresogen (1)
- moe430a (3)
- moe430b (3)
- mouse4302 (4)
- mouse430a2 (4)
- mpedbarray (1)
- mu11ksuba (3)
- mu11ksubb (3)
- Mu15v1 (1)
- mu19ksuba (2)
- mu19ksubb (2)
- mu19ksubc (2)
- Mu22v3 (1)
- mwgcod (1)
- Norway981 (1)
- OperonHumanV3 (1)
- PartheenMetaData (1)
- pedbarrayv10 (1)
- pedbarrayv9 (1)
- r10kcod (1)
- rae230a (3)
- rae230b (3)
- rat2302 (3)
- rgu34a (3)
- rgu34b (3)
- rgu34c (3)
- rgug4130a (1)
- ri16cod (1)
- rnu34 (3)
- Roberts2005Annotation (1)
- rtu34 (3)
- rwgcod (1)
- SHDZ (1)
- u133x3p (3)
- xenopuslaevis (2)
- yeast2 (3)
- ygs98 (4)
- zebrafish (3)
- CustomArray (2)
- GACustomCDF (16)
- GeneCardsCustomSchema (8)
- FunctionalAnnotation (13)
- Anopheles_gambiae (4)
- Apis_mellifera (3)
- Arabidopsis_thaliana (12)
- Bacillus_subtilis (2)
- Bos_taurus (11)
- Caenorhabditis_elegans (10)
- Canis_familiaris (12)
- Danio_rerio (12)
- Drosophila_melanogaster (15)
- Escherichia_coli (12)
- Gallus_gallus (9)
- Gasterosteus_aculeatus (2)
- Homo_sapiens (201)
- Hordeum_vulgare (2)
- Macaca_mulatta (7)
- Mus_musculus (103)
- Oryza_sativa (1)
- Pan_troglodytes (6)
- Plasmodium_falciparum (4)
- Pseudomonas_aeruginosa (2)
- Rattus_norvegicus (65)
- Saccharomyces_cerevisiae (18)
- Saccharum_officinarum (2)
- Staphylococcus_aureus (2)
- Sus_scrofa (7)
- Taeniopygia_guttata (2)
- Vitis_vinifera (2)
- Xenopus_laevis (3)
- Xenopus_tropicalis (1)
- Zea_mays (2)
- BSgenome (69)
- cdf (126)
- ChipDb (155)
- db0 (19)
- InparanoidDb (8)
- MeSHDb (3)
- OrganismDb (3)
- OrgDb (19)
- probe (104)
- SNPlocs (1)
- TxDb (17)
- miRNA (3)
- Breast (2)
- Colon (2)
- Kidney (1)
- Leukemia (6)
- Lung (1)
- Ovarian (1)
- ChIPchipData (1)
- ChIPseqData (4)
- EColiData (1)
- ExpressionData (4)
- HapMap (7)
- HighThroughputSequencingData (9)
- HIV (1)
- MassSpectrometryData (7)
- NormalTissue (3)
- RNAExpressionData (16)
- RNAseqData (19)
- StemCells (1)
- Yeast (10)
Релизы
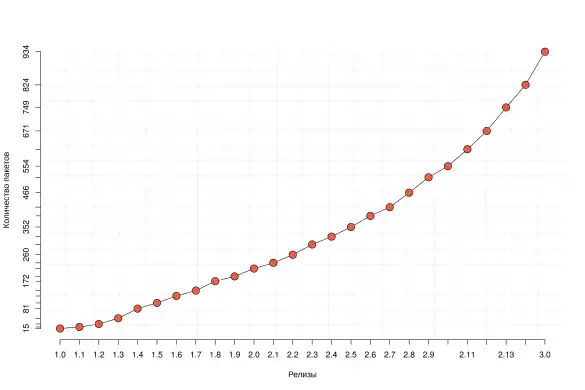
Релизы выходят два раза в год — весной и осенью. Каждый новый релиз в качестве зависимости требует последнюю версию дистрибутива R.

| Версия | Дата релиза | Количество пакетов (в разделе «Software») |
Зависимость |
|---|---|---|---|
| 1.0 | 1 мая 2001 | 15 | R 1.5 |
| 1.1 | 19 ноя 2002 | 20 | R 1.6 |
| 1.2 | 29 мая 2003 | 30 | R 1.7 |
| 1.3 | 30 окт 2003 | 49 | R 1.8 |
| 1.4 | 17 мая 2004 | 81 | R 1.9 |
| 1.5 | 25 окт 2004 | 100 | R 2.0 |
| 1.6 | 18 мая 2005 | 123 | R 2.1 |
| 1.7 | 14 окт 2005 | 141 | R 2.2 |
| 1.8 | 27 апр 2006 | 172 | R 2.3 |
| 1.9 | 4 окт 2006 | 188 | R 2.4 |
| 2.0 | 26 апр 2007 | 214 | R 2.5 |
| 2.1 | 8 окт 2007 | 233 | R 2.6 |
| 2.2 | 1 мая 2008 | 260 | R 2.7 |
| 2.3 | 22 окт 2008 | 294 | R 2.8 |
| 2.4 | 21 апр 2009 | 320 | R 2.9 |
| 2.5 | 28 окт 2009 | 352 | R 2.10 |
| 2.6 | 23 апр 2010 | 389 | R 2.11 |
| 2.7 | 18 окт 2010 | 418 | R 2.12 |
| 2.8 | 14 апр 2011 | 466 | R 2.13 |
| 2.9 | 1 ноя 2011 | 517 | R 2.14 |
| 2.10 | 2 ноя 2012 | 554 | R 2.15 |
| 2.11 | 3 окт 2012 | 610 | R 2.15 |
| 2.12 | 4 апр 2013 | 671 | R 3.0 |
| 2.13 | 15 окт 2013 | 749 | R 3.0 |
| 2.14 | 14 апр 2014 | 824 | R 3.1 |
| 3.0 | 14 окт 2014 | 934 | R 3.1 |
См. также
Примечания
- Ежегодные отчёты проекта на официальном сайте.
- R. Gentleman. [http://www.bioconductor.org/about/annual-reports/AnnRep2002.pdf 2002 Annual Report for the Bioconductor. Project, DFCI]. — May 29, 2008.
Ссылки
Официальный сайт — http://www.bioconductor.org/