Mixia osmundae
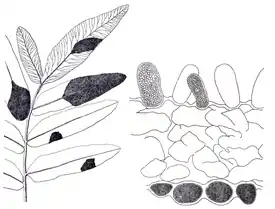
Mixia osmundae (лат.) — вид паразитических базидиальных грибов из подотдела Pucciniomycotina[3], выделяемый в монотипный класс Mixiomycetes; до 1995 года рассматривался в составе отдела сумчатых грибов (Ascomycota)[4]. Вызывают оранжевые или бурые поражения эпидермы ваий папоротников из семейства чистоустовых, хотя круг хозяев может быть и более широк[5].
| Mixia osmundae | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
 | ||||||||||
| Научная классификация | ||||||||||
| Международное научное название | ||||||||||
| Mixia osmundae (Nishida) C.L.Kramer, 1959 | ||||||||||
| Синонимы | ||||||||||
| ||||||||||
Распространение
Вид был впервые обнаружен в Японии на вайях папоротников из семейства чистоустовых — японском чистоусте (Osmunda japonica)[6]. За пределами Японии вид описан на Тайване на том же виде папоротника[2], а также на востоке США, где он отмечен на коричном чистоусте (Osmundastrum cinnamomeum) в штатах Джорджия и Мичиган[7]. Поиск в генетических базах данных выявил наличие родственного генетического материала в пробах листьев бамбуковых с территории Китая и европейского бука с территории Франции[5].
Строение в естественных условиях
На ранних стадиях заражения мицелий представляет собой тонкие ветвящиеся гифы, растущие в толще наружной клеточной стенки эпидермы хозяина, иногда заходящие в клеточные стенки мезофилла[1]. Пространство просвета гифы подразделено на сегменты простыми септами. По мере развития мицелия сегменты гиф значительно укрупняются и в результате многочисленных митотических делений ядер приобретают ценоцитное строение[1][5].
Один из исследователей вида Артур Джексон Микс отметил наличие отростков гиф к клеткам хозяина, которые он трактовал как гаустории[7], однако впоследствии факт их наличия был поставлен под сомнение[1]. Набор ферментов, закодированных в геноме Mixia osmundae, указывает, что вид, скорее всего, характеризуется биотрофным питанием[5].
Строение в условиях культуры
В культуре на питательных средах мицелий никогда не образует гиф и принимает форму дрожжей[8].
Геном
Геном Mixia osmundae был отсеквенирован в 2011 году[9]. В сравнении с геномами других исследованных фитопатогенных базидиальных грибов, он обладает наименьшим размером (13,6 млн пар оснований), высокой плотностью генов и малым количеством повторов[5].
Примечания
- Kramer C. L. A new genus in the Protomycetaceae // Mycologia. — 1958. — Vol. 50. — P. 916–926. — doi:10.2307/3755913.
- Sugiyama J., Katumoto K. Identity of the plasmodial slime mold Phytoceratiomyxa osmundae and the lectotypification of Taphrina osmundae, the basionym of Mixia osmundae // Mycoscience. — 2008. — Vol. 49, № 3. — P. 192—198. — doi:10.1007/s10267-008-0402-z.
- Bauer R., Begerow D., Sampaio J. P., Weiβ M., Oberwinkler F. The simple-septate basidiomycetes: a synopsis // Mycological Progress. — 2006. — Vol. 5, № 1. — P. 41—66. — doi:10.1007/s11557-006-0502-0.
- Nishida H., Ando K., Ando Y., Hirata A., Sugiyama J. Mixia osmundae: transfer from the Ascomycota to the Basidiomycota based on evidence from molecules and morphology. — Canadian Journal of Botany. — 1995. — Vol. 73 (suppl. 1). — P. 660—666. — doi:10.1139/b95-308.
- Toome M. et al. Genome sequencing provides insight into the reproductive biology, nutritional mode and ploidy of the fern pathogen Mixia osmundae // New Phytologist. — 2014. — Vol. 202, № 2. — P. 554—564. — doi:10.1111/nph.12653.
- Nishida T. A contribution tothe monograph of the parasitic Exoascaceae of Japan // Miyabe Festschrift. — 1911. — P. 157—212.
- Mix A. J. Taphrina osmundae Nishida and Taphrina higginsii sp. nov. // Mycologia. — 1947. — Vol. 39, № 1. — P. 71—76. — doi:10.2307/3755288.
- Nishida H., Robert V., Sugiyama J. Mixia C.L. Kramer emend. H. Nishida, K. Ando, Y. Ando, Hirata & Sugiyama (1995) // The Yeasts, a taxonomic study / Kurtzman C. P., Fell J. W., Boekhout T. (eds.). — London: Elsevier, 2011. — Vol. 3. — P. 1499—1502. — ISBN 978-0-444-52149-1. — doi:10.1016/B978-0-444-52149-1.00122-1.
- Nishida H., Nagatsuka Y., Sugiyama J. Draft genome sequencing of the enigmatic basidiomycete Mixia osmundae // Journal of General and Applied Microbiology. — Vol. 57. — P. 63—67. — doi:10.2323/jgam.57.63.